Транспортно-матрична РНК — Вікіпедія
Транспортно-матрична РНК (скорочено тмРНК, також відома як 10Sa РНК і за генетичною назвою SsrA ) — це бактеріальна молекула РНК із подвійними властивостями, подібними до транспортних та інформаційних РНК. тмРНК утворює рибонуклеопротеїновий комплекс разом із білком SmpB, фактором елонгації Tu ( EF-Tu ) і рибосомальним білком S1. тмРНК виконує ключову роль у прокаріотичному процесі транс-трансляції, необхідному для забезпечення точності трансляції білка. Коли рибосома затримуються у процесі біосинтезу білка, наприклад у випадку досягнення кінця матричної РНК, яка не містить стоп-кодону. ТмРНК виконує одразу декілька функцій: вона звільняє рибосому, що зупинилася, додає до дефектного синтезованого поліпептиду мітку, що індукує його протеоліз, та сприяє деградації аберантної інформаційної РНК . [1] У більшості бактерій ці функції виконують стандартні однокомпонентні тмРНК . В інших бактеріальних видах змінений ген ssrA виробляє двокомпонентну тмРНК, у якій два окремі ланцюги РНК з’єднані за допомогою спарювання основ.
Відкриття та початок дослідження[ред. | ред. код]
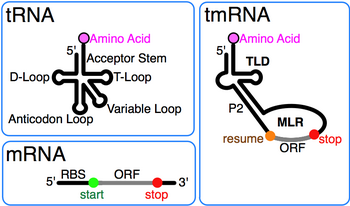
Вперше тмРНК була виділена в 1979 шляхом розділення електрофоретичної фракції 10S Escherichia coli на тмРНК (10Sa) і подібну за розміром РНК-складової РНКази Р (10Sb).[2] Присутність псевдоуридину в змішаній фракції 10S РНК вказувала на те, що тмРНК має модифіковані основи, характерні також для тРНК. При секвенуванні гену ssrA Mycobacterium tuberculosisбула виявлена подібність 3'-кінця тмРНК до TψC-петлі тРНК.[3] Подальше порівняння послідовностей виявило повний тРНК-подібний домен (TLD), утворений 5'- та 3'- кінцями тмРНК, який має акцепторну ділянку подібну до такої у аланінової тРНК, що робить можливим її аміноацилювання аланін-тРНК-синтетазою.[4] На відміну віж тРНК тмРНК не має антикодонової ділянки, а D-ділянка містить лише D-петлю без D-стебла.
Структура[ред. | ред. код]
Вторинна структура однокомпонентної тмРНК[ред. | ред. код]

Характерною особливістю усіх тмРНК є консервативний тРНК-подібний домен (TLD), що складається з спіральних ділянок 1, 12 і 2a (аналогів акцепторного, Т- і варіабельного стебел тРНК відповідно) і містить 5' монофосфатний і аланільований 3' CCA-кінець. мРНК-подібна ділянка (MLR) у стандартній тмРНК є великою петлею, що містить псевдовузли і кодуючу послідовність (CDS) для пептидної мітки довжиною 10-27 амінокислот, обмежену кодоном відновлення і стоп-кодоном. Кодований пептид мітки варіює між бактеріями, можливо, залежно від набору наявних протеаз та адаптерів.[5]
тмРНК зазвичай містять чотири псевдовузли, один з яких (pk1) знаходиться вище ділянки CDS, а інші три псевдовузли (pk2 - pk4) - нижче за CDS. Попри загальну косервативність тмРНК, псевдовузли є достатньо еволюційно пластичними. Наприклад, у однокомпонентних тмРНК ціанобактерій pk4 заміщений двома тандемно розташованими меншими псевдовузлами. Це свідчить про те, що вторинна структура тмРНК за межами TLD є важливою, проте псевдовузли є одними з перших структур, які втрачаються при розбіжності послідовностей ssrA в лініях пластид та інших ендосимбіонтів.[6][7]
Двокомпонентні тмРНК[ред. | ред. код]
Кругова пермутація[en] ssrA була виявлена в трьох основних лініях:
- у всіх альфа-протеобактерій і примітивних мітохондріях протистів Jakobida;
- у двох розрізнених групах ціанобактерій (Gloeobacter і клада, що містить Prochlorococcus і багато представників Synechococcus);
- у деяких представників бета-протеобактерій (Cupriavidus і деяких Rhodocyclales).[8][9]
Всі вони утворюють подібні двокомпонентні мтРНК, що складаються з акцепторної та кодуючої частин. Така мтРНК еквівалентна стандартній формі, але розрізана після кодувальної послідовності CDS. У таких мтРНК зберігається не більше 2 псевдовузлів, порівняно з 4 чи більше в стандартних мтРНК.
Альфапротеобактерії мають дві характерні послідовності: заміну типової послідовності ТΨC-петлі TΨCRANY на GGCRGUA та послідовність AACAGAA у великій петлі 3´-кінцевого псевдовузла. У мітохондріях MLR була втрачена. Значна репермутація мітохондріальної ssrA призводить до утворення невеликого однокомпонентного продукту у Jakoba libera.[10]
Ціанобактерії є найбільш вірогідним прикладом утворення пермутованого гена зі стандартного, через значну схожість послідовностей обох типів генів, які зустрічаються в різних штамах.
Процесинг тмРНК[ред. | ред. код]
Більшість тмРНК транскрибуються у вигляді більших попередників, які піддаються процесингу подібно до тРНК. Розщеплення на 5'-кінці відбувається за допомогою рибонуклеази Р.[11] 3'-кінець найчастіше піддається процесингу за допомогою РНКаз Т та PH.[12][13] Залежно від виду бактерій, 3'-CCA або кодується, або додається за допомогою тРНК-нуклеотидилтрансферази.
Подібний процесинг у внутрішніх сайтах попередниці тмРНК пояснює її фізичне розщеплення на дві частини. Двокомпонентні тмРНК мають два додаткові кінці, процесинг яких необхідно враховувати. Для альфапротеобактерій один 5'-кінець є необробленим сайтом початку транскрипції. [14] Дальній 3'-кінець може в деяких випадках бути результатом rho-незалежної термінації транскрипції.
Тривимірні конструкції[ред. | ред. код]


Структурні моделі з високою роздільною здатністю повних молекул тмРНК наразі недоступні, і їх може бути важко отримати через притаманну MLR гнучкість. У 2007 році була отримана кристалічна структура тмРНК Thermus thermophilus, зв'язаної з білком SmpB, з роздільною здатністю 3 Å. Ця структура показує, що SmpB імітує D-стебло та антикодон канонічної тРНК, тоді як спіральна ділянка 2a тмРНК відповідає варіабельному плечу тРНК.[17] Кріоелектронне мікроскопічне дослідження тмРНК на ранній стадії транс-трансляції показує просторові взаємовідносини між рибосомою і тмРНК (тмРНК, зв'язаною з білком EF-Tu). TLD розташований поблизу ГТФазо-асоційованого центру в 50S субодиниці рибосоми; спіральна область 5 і псевдовузли pk2 - pk4 утворюють дугу навколо 30S субодиниці рибосоми.[18]
Транс-трансляція[ред. | ред. код]

Кодування тмРНК було відкрито в 1995 році, коли Сімпсон з колегами оверекспресували рекомбінантний мишачий цитокін IL-6 в E. coli і виявили кілька усічених пептидів, похідних від цитокінів, кожен з яких на С-кінцях мав однаковий 11-амінокислотного залишку (A)ANDENYALAA.[19] За винятком N-кінцевого аланіну, який походить з 3' кінця самої тмРНК, ця послідовність походить від короткої відкритої рамки зчитування тмРНК E. coli. Кейлер та ін. з'ясували, що така пептидна мітка забезпечує протеоліз, і запропонували модель транс-трансляції, опосередкованої тмРНК.[20]
Хоча деталі механізму транс-трансляції ще досліджуються, загальновизнано, що тмРНК спочатку зв'язується з ділянкою А рибосоми, що зупинилася у процесі трансляції. Згодом рибосома переміщується з 3' кінця пошкодженої мРНК на кодон відновлення MLR, після чого трансляція продовжується в нормальному режимі на матриці кодуючої послідовності тмРНК, поки не доходить до стоп-кодону. Деяким видам бактерій транс-трансляція життєво необхідна, тоді як інші використовують її лише у стресових умовах.[21] Вважається, що тмРНК може відігравати роль у антибіотикорезистентності, рятуючи рибосоми, зупинені антибіотиками, що впливають на трансляцію.[22] Залежно від організму, пептидні мітки можуть розпізнаватися різноманітними протеазами або протеазними адаптерами.[23]
Вплив мобільних генетичних елементів на ген тмРНК[ред. | ред. код]
ssrA може як бути мішенню для одних мобільних генетичних елементів, так і переноситись за допомогою інших МГЕ. Було виявлено встроювання трьох типів МГЕ у цей ген. Жоден з них не порушує функцію гена: інтрони групи I видаляються шляхом самосплайсингу, рикетсійні паліндромні елементи (RPE) вбудовуються в некритичні ділянки, а геномні острівці, що кодують інтегразу, роз'єднують ssrA, але відновлюють одну з відщеплених ділянок.[24][25][26][27]
Нехромосомний ssrA був вперше виявлений в геномному дослідженні мікобактеріофагів (у 10% фагів).[28] Також були знайдені інші МГЕ, що містять ssrA, наприклад, плазміди та геномні острівці. Одним з цікавих випадків є Rhodobacter sphaeroides ATCC 17025, чий нативний ген тмРНК порушений геномним острівцем. На відміну від усіх інших геномних острівців у генах тмРНК (або тРНК), цей острів інактивував нативний ген-мішень без відновлення відщепленого фрагмента, але компенсував це за рахунок наявності власного гена тмРНК. Дуже незвичайний родич ssrA виявлений у літичного мікобактеріофага DS6A, який кодує трохи більше, ніж TLD.
Мітохондріальні тмРНК[ред. | ред. код]
Мітохондріальна, структурно редукована форма тмРНК (мт-тмРНК) була вперше виявлена у джгутикових найпростіших Reclinomonas americana.[29] Згодом наявність мітохондріального гена ssrA, що кодує тмРНК, а також сайтів транскрипції та процесингу РНК було підтверджено для всіх представників якобід, окрім одного.[30][31] Докази функціональності - аміноацилювання мт-тмРНК аланіном - виявлені у Jakoba libera.[31] Нещодавно ssrA також було ідентифіковано в мітохондріальних геномах ооміцетів.[32] Як і в α-протеобактерій (предків мітохондрій), mt-тмРНК мають кругову пермутацію, тобто представлені двокомпонентними молекулами, за винятком Jakoba libera, де ген повернувся до кодування однокомпонентної тмРНК.[31]
Ідентифікація ssrA в мітохондріальних геномах[ред. | ред. код]

Спочатку гени мітохондріальної тмРНК визначались за короткими послідовностями, консервативними серед якобід, що схильні згортатись у тРНК-подібну вторинну структуру. Завдяки наявності дев'яти повних послідовностей мтДНК якобід[33] та значному вдосконаленню інструментів пошуку коваріацій (Infernal;[34][35][36]) було розроблено коваріаційну модель на основі мітохондріальних тмРНК якобіда, за допомогою якої ідентифіковано гени мітохондріальної ssrA у ооміцетів. Наразі виявлено 34 мт-тмРНК ооміцетів у шести родах: Albugo, Bremia, Phytophthora, Pseudoperonospora, Pythium та Saprolegnia. Коваріаційна модель, побудована з використанням послідовностей якобід і ооміцетів, тепер доступна на Rfam під назвою "mt-tmRNA".[37]
Структура мт-тмРНК[ред. | ред. код]
Стандартна бактеріальна тмРНК складається з тРНК(Ala)-подібного домену (дозволяє додавати некодований аланін до мРНК, які не мають стоп-коду) та мРНК-подібного домену, що кодує білкову мітку, яка спрямовує поліпептид на протеоліз. мРНК-подібний домен був втрачений у мт-тмРНК. Порівняльний аналіз послідовностей вказує на особливості, характерні для мт-тмРНК.[38] Найбільш консервативною є первинна послідовність аміноацил-акцепторної основи. Ця частина молекули має незмінний залишок А в позиції дискримінатора і пару G-U в позиції 3 (за винятком Seculamonas ecuadoriensis, яка має пару G-C). Ця позиція є сайтом впізнавання для аланіл-тРНК-синтази.
P2 - це спіраль змінної довжини (від 3 до 10 пар основ), що відповідає антикодоновій ділянці тРНК, але без антикодонової петлі (оскільки вона не потрібна для функціонування тмРНК). P2 стабілізує тРНК-подібну структуру, але чотири нуклеотиди, інваріантні для ооміцетів та якобід, вказують на додаткову, наразі не ідентифіковану функцію. P3 має п'ять пар основ і відповідає ТψC-ділянці тРНК, але з різними консенсусними нуклеотидами як у стеблі, так і в петлі. Послідовність ТψC-петлі консервативна у ооміцетів та якобід, з невеликими відхиленнями (наприклад, у Saprolegnia ferax). Нарешті, замість тРНК-подібної D-ділянки з укороченою тринуклеотидною D-петлею, характерної для бактеріальних тмРНК, мітохондріальні аналоги мають дуже варіабельну петлю довжиною від 5 до 14 п.н.

Дані секвенування РНК Phytophthora sojae показують рівень експресії мт-тмРНК, подібний до рівня експресії сусідніх мітохондріальних тРНК, а чотири основні процесингові сайти підтверджують передбачувані кінцеві ділянки зрілої мт-тмРНК.[39] Молекула-попередник тмРНК, ймовірно, обробляється РНКазою Р та специфічною ендонуклеазою, що забезпечує процесинг 3' тмРНК. Остання, як припускають, видаляє проміжну послідовность. Після додавання CCA до 3' дискримінаторного нуклеотиду, до тмРНК приєднанується залишок аланіну за допомогою аланіл-тРНК-синтетази.
Див. також[ред. | ред. код]
Примітки[ред. | ред. код]
- ↑ Keiler KC (2008). Biology of trans-translation. Annual Review of Microbiology. 62: 133—51. doi:10.1146/annurev.micro.62.081307.162948. PMID 18557701.
- ↑ Ray BK, Apirion D (July 1979). Characterization of 10S RNA: a new stable rna molecule from Escherichia coli. Molecular & General Genetics. 174 (1): 25—32. doi:10.1007/BF00433301. PMID 384159.
- ↑ Tyagi JS, Kinger AK (January 1992). Identification of the 10Sa RNA structural gene of Mycobacterium tuberculosis. Nucleic Acids Research. 20 (1): 138. doi:10.1093/nar/20.1.138. PMC 310338. PMID 1371186.
- ↑ Komine Y, Kitabatake M, Yokogawa T, Nishikawa K, Inokuchi H (September 1994). A tRNA-like structure is present in 10Sa RNA, a small stable RNA from Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 91 (20): 9223—7. doi:10.1073/pnas.91.20.9223. PMC 44784. PMID 7524073.
- ↑ Gur E, Sauer RT (October 2008). Evolution of the ssrA degradation tag in Mycoplasma: specificity switch to a different protease. Proceedings of the National Academy of Sciences of the United States of America. 105 (42): 16113—8. doi:10.1073/pnas.0808802105. PMC 2570983. PMID 18852454.
- ↑ Zwieb C, Wower I, Wower J (May 1999). Comparative sequence analysis of tmRNA. Nucleic Acids Research. 27 (10): 2063—71. doi:10.1093/nar/27.10.2063. PMC 148424. PMID 10219077.
- ↑ Wower IK, Zwieb C, Wower J (May 2005). Transfer-messenger RNA unfolds as it transits the ribosome. RNA. 11 (5): 668—73. doi:10.1261/rna.7269305. PMC 1370753. PMID 15811920.
- ↑ Keiler KC, Shapiro L, Williams KP (July 2000). tmRNAs that encode proteolysis-inducing tags are found in all known bacterial genomes: A two-piece tmRNA functions in Caulobacter. Proceedings of the National Academy of Sciences of the United States of America. 97 (14): 7778—83. doi:10.1073/pnas.97.14.7778. PMC 16621. PMID 10884408.
- ↑ Sharkady SM, Williams KP (2004). A third lineage with two-piece tmRNA. Nucleic Acids Research. 32 (15): 4531—8. doi:10.1093/nar/gkh795. PMC 516066. PMID 15326226.
- ↑ Jacob Y, Seif E, Paquet PO, Lang BF (April 2004). Loss of the mRNA-like region in mitochondrial tmRNAs of jakobids. RNA. 10 (4): 605—14. doi:10.1261/rna.5227904. PMC 1370551. PMID 15037770.
- ↑ Komine Y, Kitabatake M, Yokogawa T, Nishikawa K, Inokuchi H (September 1994). A tRNA-like structure is present in 10Sa RNA, a small stable RNA from Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 91 (20): 9223—7. doi:10.1073/pnas.91.20.9223. PMC 44784. PMID 7524073.
- ↑ Srivastava RA, Srivastava N, Apirion D (May 1992). Characterization of the RNA processing enzyme RNase III from wild type and overexpressing Escherichia coli cells in processing natural RNA substrates. The International Journal of Biochemistry. 24 (5): 737—49. doi:10.1016/0020-711X(92)90007-N. PMID 1375563.
- ↑ Li Z, Pandit S, Deutscher MP (March 1998). 3' exoribonucleolytic trimming is a common feature of the maturation of small, stable RNAs in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 95 (6): 2856—61. doi:10.1073/pnas.95.6.2856. PMC 19659. PMID 9501180.
- ↑ Mao C, Bhardwaj K, Sharkady SM, Fish RI, Driscoll T, Wower J, Zwieb C, Sobral BW, Williams KP (2009). Variations on the tmRNA gene. RNA Biology. 6 (4): 355—61. doi:10.4161/rna.6.4.9172. PMID 19617710.
- ↑ Someya T, Nameki N, Hosoi H, Suzuki S, Hatanaka H, Fujii M, Terada T, Shirouzu M, Inoue Y, Shibata T, Kuramitsu S, Yokoyama S, Kawai G (January 2003). Solution structure of a tmRNA-binding protein, SmpB, from Thermus thermophilus. FEBS Letters. 535 (1–3): 94—100. doi:10.1016/S0014-5793(02)03880-2. PMID 12560085.
- ↑ Bessho Y, Shibata R, Sekine S, Murayama K, Higashijima K, Hori-Takemoto C, Shirouzu M, Kuramitsu S, Yokoyama S (May 2007). Structural basis for functional mimicry of long-variable-arm tRNA by transfer-messenger RNA. Proceedings of the National Academy of Sciences of the United States of America. 104 (20): 8293—8. doi:10.1073/pnas.0700402104. PMC 1895943. PMID 17488812.
- ↑ Bessho Y, Shibata R, Sekine S, Murayama K, Higashijima K, Hori-Takemoto C, Shirouzu M, Kuramitsu S, Yokoyama S (May 2007). Structural basis for functional mimicry of long-variable-arm tRNA by transfer-messenger RNA. Proceedings of the National Academy of Sciences of the United States of America. 104 (20): 8293—8. doi:10.1073/pnas.0700402104. PMC 1895943. PMID 17488812.
- ↑ Valle M, Gillet R, Kaur S, Henne A, Ramakrishnan V, Frank J (April 2003). Visualizing tmRNA entry into a stalled ribosome. Science. 300 (5616): 127—30. doi:10.1126/science.1081798. PMID 12677067.
- ↑ Tu GF, Reid GE, Zhang JG, Moritz RL, Simpson RJ (April 1995). C-terminal extension of truncated recombinant proteins in Escherichia coli with a 10Sa RNA decapeptide. The Journal of Biological Chemistry. 270 (16): 9322—6. doi:10.1074/jbc.270.16.9322. PMID 7536743.
- ↑ Keiler KC, Waller PR, Sauer RT (February 1996). Role of a peptide tagging system in degradation of proteins synthesized from damaged messenger RNA. Science. 271 (5251): 990—3. doi:10.1126/science.271.5251.990. PMID 8584937.
- ↑ Thibonnier M, Thiberge JM, De Reuse H (2008). Ahmed, Niyaz (ред.). Trans-translation in Helicobacter pylori: essentiality of ribosome rescue and requirement of protein tagging for stress resistance and competence. PLOS ONE. 3 (11): e3810. doi:10.1371/journal.pone.0003810. PMC 2584231. PMID 19043582.
- ↑ Müller, Claudia; Crowe-McAuliffe, Caillan; Wilson, Daniel N. (18 березня 2021). Ribosome Rescue Pathways in Bacteria. Frontiers in Microbiology. 12: 652980. doi:10.3389/fmicb.2021.652980. ISSN 1664-302X. PMC 8012679. PMID 33815344.
- ↑ Gur E, Sauer RT (October 2008). Evolution of the ssrA degradation tag in Mycoplasma: specificity switch to a different protease. Proceedings of the National Academy of Sciences of the United States of America. 105 (42): 16113—8. doi:10.1073/pnas.0808802105. PMC 2570983. PMID 18852454.
- ↑ Kirby JE, Trempy JE, Gottesman S (April 1994). Excision of a P4-like cryptic prophage leads to Alp protease expression in Escherichia coli. Journal of Bacteriology. 176 (7): 2068—81. doi:10.1128/jb.176.7.2068-2081.1994. PMC 205313. PMID 7511583.
- ↑ Williams KP (January 2002). The tmRNA Website: invasion by an intron. Nucleic Acids Research. 30 (1): 179—82. doi:10.1093/nar/30.1.179. PMC 99078. PMID 11752287.
- ↑ Dwyer DS (January 2001). Selfish DNA and the origin of genes. Science. 291 (5502): 252—3. doi:10.1126/science.291.5502.252. PMID 11253208.
- ↑ Williams KP (February 2003). Traffic at the tmRNA gene. Journal of Bacteriology. 185 (3): 1059—70. doi:10.1128/JB.185.3.1059-1070.2003. PMC 142792. PMID 12533482.
- ↑ Hatfull GF, Pedulla ML, Jacobs-Sera D, Cichon PM, Foley A, Ford ME, Gonda RM, Houtz JM, Hryckowian AJ, Kelchner VA, Namburi S, Pajcini KV, Popovich MG, Schleicher DT, Simanek BZ, Smith AL, Zdanowicz GM, Kumar V, Peebles CL, Jacobs WR, Lawrence JG, Hendrix RW (June 2006). Exploring the mycobacteriophage metaproteome: phage genomics as an educational platform. PLOS Genetics. 2 (6): e92. doi:10.1371/journal.pgen.0020092. PMC 1475703. PMID 16789831.
{{cite journal}}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Keiler KC, Shapiro L, Williams KP (July 2000). tmRNAs that encode proteolysis-inducing tags are found in all known bacterial genomes: A two-piece tmRNA functions in Caulobacter. Proceedings of the National Academy of Sciences of the United States of America. 97 (14): 7778—83. doi:10.1073/pnas.97.14.7778. PMC 16621. PMID 10884408.
- ↑ Burger G, Gray MW, Forget L, Lang BF (2013). Strikingly bacteria-like and gene-rich mitochondrial genomes throughout jakobid protists. Genome Biology and Evolution. 5 (2): 418—38. doi:10.1093/gbe/evt008. PMC 3590771. PMID 23335123.
- ↑ а б в Jacob Y, Seif E, Paquet PO, Lang BF (April 2004). Loss of the mRNA-like region in mitochondrial tmRNAs of jakobids. RNA. 10 (4): 605—14. doi:10.1261/rna.5227904. PMC 1370551. PMID 15037770.
- ↑ Hafez M, Burger G, Steinberg SV, Lang BF (July 2013). A second eukaryotic group with mitochondrion-encoded tmRNA: in silico identification and experimental confirmation. RNA Biology. 10 (7): 1117—24. doi:10.4161/rna.25376. PMC 3849159. PMID 23823571. Архів оригіналу за 21 лютого 2014. Процитовано 13 лютого 2014.
- ↑ Burger G, Gray MW, Forget L, Lang BF (2013). Strikingly bacteria-like and gene-rich mitochondrial genomes throughout jakobid protists. Genome Biology and Evolution. 5 (2): 418—38. doi:10.1093/gbe/evt008. PMC 3590771. PMID 23335123.
- ↑ Eddy, S. Infernal website. Процитовано 14 August 2016.
- ↑ Eddy SR, Durbin R (June 1994). RNA sequence analysis using covariance models. Nucleic Acids Research. 22 (11): 2079—88. doi:10.1093/nar/22.11.2079. PMC 308124. PMID 8029015.
- ↑ Nawrocki EP, Kolbe DL, Eddy SR (May 2009). Infernal 1.0: inference of RNA alignments. Bioinformatics. 25 (10): 1335—7. doi:10.1093/bioinformatics/btp157. PMC 2732312. PMID 19307242.
- ↑ Hafez M, Burger G, Steinberg SV, Lang BF (July 2013). A second eukaryotic group with mitochondrion-encoded tmRNA: in silico identification and experimental confirmation. RNA Biology. 10 (7): 1117—24. doi:10.4161/rna.25376. PMC 3849159. PMID 23823571. Архів оригіналу за 21 лютого 2014. Процитовано 13 лютого 2014.
- ↑ Hafez M, Burger G, Steinberg SV, Lang BF (July 2013). A second eukaryotic group with mitochondrion-encoded tmRNA: in silico identification and experimental confirmation. RNA Biology. 10 (7): 1117—24. doi:10.4161/rna.25376. PMC 3849159. PMID 23823571. Архів оригіналу за 21 лютого 2014. Процитовано 13 лютого 2014.
- ↑ Hafez M, Burger G, Steinberg SV, Lang BF (July 2013). A second eukaryotic group with mitochondrion-encoded tmRNA: in silico identification and experimental confirmation. RNA Biology. 10 (7): 1117—24. doi:10.4161/rna.25376. PMC 3849159. PMID 23823571. Архів оригіналу за 21 лютого 2014. Процитовано 13 лютого 2014.
| ||||||||||||||||||||


 French
French Deutsch
Deutsch