Helgenomstudie – Wikipedia

En helgenomstudie (engelska: Genome-Wide Association Study, GWAS) är en studiemetod inom genomiken. Metoden är en form av observationsstudie där man går igenom genvarianter hos flera individer för att se vilka som är kopplade till en viss fenotyp. Syftet med studierna är vanligen att påvisa korrelation mellan enbaspolymorfier (engelska: SNP för Single Nucleotide Polymorphy) och sjukdomsförekomst hos människan. Metoden kan dock tillämpas på valfri organism och kan studera valfri fenotyp som regleras via genuttryck, inte bara sjukdomar. Helgenomstudier undersöker hela genomet, i motsats till olika andra metoder som specifikt testar ett litet antal förutbestämda genetiska regioner. Helgenomstudier är därför inte beroende av att man på förhand identifierar och väljer ut ett antal kandidatgener att studera.
Inom humanmedicin jämför helgenomstudier arvsmassan hos studiedeltagare med olika fenotyper för ett visst drag, vanligen en sjukdom. Dessa deltagare delas då upp i olika grupper där den ena gruppen är drabbade (fall) och den andra är friska (kontroller). Varje person ger ett urval av DNA, från vilket miljoner genetiska varianter läses med SNP-matriser. Om en genvariant är vanligare hos personer med sjukdomen, sägs varianten vara associerad till sjukdomen. Helgenomstudier kan inte på egen hand bevisa orsakssamband.[1][2][3] Den första framgångsrika GWAS publicerades 2002 och studerade hjärtinfarkt. Studiedesignen återanvändes 2005 i en studie av åldersrelaterad makuladegeneration där man kunde identifiera två SNP med signifikant förändrad genfrekvens jämfört med friska kontroller. År 2017 hade över 3000 helgenomstudier genomförts på människor med totalt över 1 800 sjukdomar eller andra egenskaper undersökta och identifierat tusentals associerade SNP som följd.
Bakgrund[redigera | redigera wikitext]
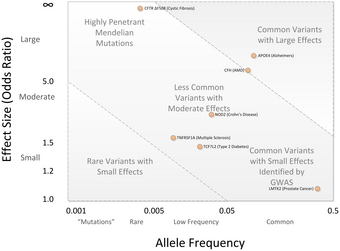
Två slumpvis valda mänskliga genom skiljer sig åt på flera miljoner baspar. Det finns variationer i de enskilda nukleotiderna i genomen (SNP) såväl som större skillnader på flera baspar i rad. Båda dessa kan orsaka förändringar hos en persons egenskaper, till exempel sjukdomsrisk eller fysiska egenskaper såsom längd.[5] Omkring år 2000, före införandet av GWA-studier, var den primära undersökningsmetoden genom arvstudier av genetisk koppling i familjer. Detta tillvägagångssätt hade visat sig vara mycket användbart mot enstaka genstörningar.[6][5][7] Men för vanliga och komplexa sjukdomar visade sig resultaten av genetiska kopplingsstudier svåra att reproducera.[5][7] Ett föreslaget alternativ till kopplingsstudier var den genetiska associeringsstudien. Denna studietyp frågar om allelen av en genetisk variant hittas oftare än väntat hos individer med fenotypen av intresse (t.ex. med den sjukdom som studeras). Tidiga beräkningar av statistisk effekt indikerade att detta tillvägagångssätt kunde vara bättre än kopplingsstudier för att upptäcka svaga genetiska effekter.[8]
Flera faktorer underlättade genomförandet av de första helgenomstudierna. En var tillkomsten av biobanker, som underlättade insamlandet av de stora mängder provmaterial som krävdes för att genomföra studerna [9] En annan var International HapMap Project som hjälpte till att identifiera majoriteten av de SNP som undersöks i helgenomstudier.[10]
Den flesta helgenomstudier är designade som fall-kontrollstuder. Två stora grupper individer jämförs med varandra, en frisk kontrollgrupp och en fallgrupp är drabbade av en viss sjukdom. Alla individer i grupperna genotypas för de flesta vanliga och kända SNP. Det exakta antalet SNP beror på metoden för genotypning, men är vanligtvis en miljon och uppåt.[4] För var och en av dessa SNP undersöks det sedan om allelfrekvensen ändras signifikant mellan fallgrupp och kontrollgrupp.[11] Effektstorleken mellan de olika grupperna återges som en oddskvot.
Försök har gjorts att skapa kataloger över SNP som har identifierats i helgenomstudier.[12] Redan 2009 fanns tusentals kända SNP som kopplats till ökad risk för sjukdom.[13]
Referenser[redigera | redigera wikitext]
- Den här artikeln är helt eller delvis baserad på material från engelskspråkiga Wikipedia, 14 augusti 2021.
- ^ Manolio, Teri A. (2010-07-08). W. Gregory Feero, Alan E. Guttmacher. red. ”Genomewide Association Studies and Assessment of the Risk of Disease” (på engelska). New England Journal of Medicine 363 (2): sid. 166–176. doi:. ISSN 0028-4793. http://www.nejm.org/doi/10.1056/NEJMra0905980. Läst 2 november 2021.
- ^ Pearson, Thomas A.; Manolio, Teri A. (2008-03-19). ”How to interpret a genome-wide association study”. JAMA 299 (11): sid. 1335–1344. doi:. ISSN 1538-3598. PMID 18349094. https://pubmed.ncbi.nlm.nih.gov/18349094. Läst 2 november 2021.
- ^ ”Genome-Wide Association Studies”. Genome-Wide Association Studies. National Human Genome Research Institute. http://www.genome.gov/20019523.
- ^ [a b] ”Chapter 11: Genome-wide association studies”. PLOS Computational Biology 8 (12): sid. e1002822. 2012. doi:. PMID 23300413. Bibcode: 2012PLSCB...8E2822B.
- ^ [a b c] Human Molecular Genetics. Garland Science. https://archive.org/details/humanmolecularge00stra_254.
- ^ ”Online Mendelian Inheritance in Man”. Online Mendelian Inheritance in Man. http://www.omim.org.
- ^ [a b] Altmüller, Janine; Palmer, Lyle J.; Fischer, Guido; Scherb, Hagen; Wjst, Matthias (2001-11). ”Genomewide Scans of Complex Human Diseases: True Linkage Is Hard to Find” (på engelska). The American Journal of Human Genetics 69 (5): sid. 936–950. doi:. https://linkinghub.elsevier.com/retrieve/pii/S000292970761310X. Läst 2 november 2021.
- ^ Risch, N.; Merikangas, K. (1996-09-13). ”The future of genetic studies of complex human diseases”. Science (New York, N.Y.) 273 (5281): sid. 1516–1517. doi:. ISSN 0036-8075. PMID 8801636. https://pubmed.ncbi.nlm.nih.gov/8801636. Läst 2 november 2021.
- ^ ”The uneasy ethical and legal underpinnings of large-scale genomic biobanks”. Annual Review of Genomics and Human Genetics 8: sid. 343–64. 2007. doi:. PMID 17550341.
- ^ ”The International HapMap Project”. Nature 426: sid. 789–96. December 2003. doi:. PMID 14685227.
- ^ ”Basic statistical analysis in genetic case-control studies”. Nature Protocols 6 (2): sid. 121–33. February 2011. doi:. PMID 21293453.
- ^ ”Potential etiologic and functional implications of genome-wide association loci for human diseases and traits”. Proceedings of the National Academy of Sciences of the United States of America 106 (23): sid. 9362–7. June 2009. doi:. PMID 19474294. Bibcode: 2009PNAS..106.9362H.
- ^ ”An open access database of genome-wide association results”. BMC Medical Genetics 10: sid. 6. January 2009. doi:. PMID 19161620.
Externa länkar[redigera | redigera wikitext]
- Whole genome association studies av National Human Genome Research Institute


 French
French Deutsch
Deutsch