Гомологичная рекомбинация — Википедия

Гомологи́чная рекомбина́ция, или о́бщая рекомбина́ция[1], — тип генетической рекомбинации, во время которой происходит обмен нуклеотидными последовательностями между двумя похожими или идентичными хромосомами. Это наиболее широко используемый клетками способ устранения двух- или однонитевых повреждений ДНК. Гомологичная рекомбинация также создаёт разнообразие комбинаций генов во время мейоза, обеспечивающих высокий уровень наследственной изменчивости, что, в свою очередь, позволяет популяции лучше адаптироваться в ходе эволюции[1]. Различные штаммы и виды бактерий и вирусов используют гомологичную рекомбинацию в процессе горизонтального переноса генов.
Хотя механизм гомологичной рекомбинации (ГР) широко варьируется среди разных организмов и типов клеток, зачастую в её основе лежит один и тот же механизм. Разрыв двух нитей ДНК приводит к тому, что 5'-концы непосредственно рядом с повреждением удаляются. Следующий шаг заключается во внедрении или инвазии 3'-конца повреждённой цепи в другую, целую ДНК, используемую в качестве матрицы. Дальнейшая последовательность событий может идти двумя путями (описанными ниже), известными как DSBR или SDSA. Гомологическая рекомбинация, происходящая во время репарации ДНК, как правило, приводит к восстановлению молекулы в том же виде, в котором та находилась до повреждения.
Поскольку явление ГР прослеживается во всех трёх доменах живой природы, а также у вирусов, его можно считать универсальным биологическим механизмом. Открытие ГР-генов у протистов — разнообразной группы эукариотических микроорганизмов — было истолковано как свидетельство того, что мейоз возник на раннем этапе эволюции эукариот. Так как нарушение работы этих генов часто связана с возникновением нескольких видов рака, белки, кодируемые этими генами и участвующие в процессе ГР, являются предметом активных исследований. На гомологической рекомбинации построен также генетический таргетинг — процесс, при котором в геном организма вносятся искусственные изменения. За развитие этой технологии Марио Капекки, Мартин Эванс и Оливер Смитис были удостоены в 2007 году Нобелевской премии по физиологии и медицине. Капекки[2] и Смитис[3] независимо друг от друга открыли способ редактирования геномов эмбриональных стволовых клеток мышей, однако, высококонсервативные механизмы, лежащие в основе репарации повреждений ДНК, в том числе вставки изменённой последовательности гена при генной терапии, были впервые изучены в экспериментах с плазмидами, проведённых Терри Орр-Вивер, Джеком Шостаком и Родни Ротштейном[4][5][6]. Исследование плазмид, облучённых γ-излучением[7], привело к экспериментам, в ходе которых при помощи эндонуклеаз разрезались хромосомы для нужд генной инженерии клеток млекопитающих, где негомологичная рекомбинация встречается чаще, чем у дрожжей[8].
История изучения[править | править код]

В начале 1900-х годов Уильям Бейтсон и Реджинальд Паннет нашли исключение из одного из законов Менделя, первоначально описанных Грегором Менделем в 1860-х годах. В отличие от идеи Менделя о том, что признаки наследуются независимо друг от друга при передаче их потомству, Бейтсон и Паннет показали, что некоторые гены, связанные с физическими признаками, могут быть унаследованы вместе, или генетически связаны[9][10]. В 1911 году, когда выяснилось, что связанные черты могут иногда передаваться отдельно, Томас Хант Морган предположил, что между связанными генами происходит кроссинговер[11], во время которого один из сцепленных генов физически переходит на другую хромосому. Двадцать лет спустя Барбара Мак-Клинток и Харриет Крейтон доказали, что обмен участками хромосом происходит при мейозе[12][13], обычно связанном с образованием гамет. В тот же год, когда было совершено открытие Мак-Клинток, Курт Штерн показал, что кроссинговер может также происходить в соматических клетках, таких как лейкоциты и клетки кожи, которые делятся митозом[12][14].
В 1947 году микробиолог Джошуа Ледерберг продемонстрировал, что бактерии, которые, как предполагалось, размножаются только путём бинарного деления, обладают способностью к генетической рекомбинации, которая больше напоминает половое размножение. Эта работа основывалась на исследовании кишечной палочки Escherichia coli[15], и за неё Ледерберг был удостоен Нобелевской премии по физиологии и медицине в 1958 году[16]. В 1964 году, опираясь на исследования грибов, Робин Холлидей предложил модель рекомбинации в мейозе, которая включала в себя все ключевые аспекты этого процесса, в том числе обмен генетическим материалом между хромосомами посредством образования структуры Холлидея[17]. В 1983 году Джек Шостак и его коллеги представили другую модель, известную сейчас как путь репарации двуцепочечных разрывов (англ. double-strand break repair, DSBR), толковавшую те детали, которые не смогла объяснить модель Холлидея[17][6]. В течение следующих десяти лет в результате экспериментов на дрозофилах, дрожжах и клетках млекопитающих был обнаружен ещё один тип гомологической рекомбинации, не всегда следующий модели Холлидея, который был назван путём синтез-зависимого отжига цепей (англ. synthesis-dependent strand annealing, SDSA)[17].
У эукариот[править | править код]
Гомологичная рекомбинация имеет важное значение в делении клеток эукариот: растений, животных, грибов и протистов. В клетках, делящихся путём митоза, ГР является инструментом репарации повреждений ДНК, вызванных ионизирующим излучением или химическими веществами[18]. Будучи неотрепарированными, эти повреждения способны привести к масштабной перестройке хромосом в соматических клетках[19], а потом и к раку[20].
В дополнение к устранению повреждений, ГР обеспечивает генетическое разнообразие при мейотическом делении с последующим образованием гамет и спор. Центральную роль здесь играет кроссинговер, в результате которого хромосомы обмениваются участками ДНК[21][22]. Благодаря этому появляются новые, возможно, полезные комбинации генов, которые могут дать потомству эволюционное преимущество[1]. Чаще всего кроссинговер начинается, когда белок Spo11 делает целевые двойные разрезы в цепи ДНК[23] строго определённым образом, преимущественно в промоторах и GC-обогащённых областях[24]. Обычно эти области находятся в так называемых горячих точках рекомбинации — участках, состоящих из приблизительно 1000—2000 пар оснований и имеющих высокую частоту рекомбинации. Отсутствие горячих точек рядом с двумя генами в одной и той же хромосоме часто означает, что эти гены будут унаследованы будущими поколениями в равной пропорции[25].
Регуляция[править | править код]

Двойные разрывы ДНК-цепи могут быть исправлены либо путём гомологичной рекомбинации, либо негомологичным соединением концов (НСК, англ. non-homologous end joining, NHEJ). НСК (NHEJ) — механизм репарации, который, в отличие от ГР, не требует гомологичной матрицы. Выбор одного из этих двух механизмов репарации во многом определяется фазой клеточного цикла. ГР возможна во время S-фазы и G2-стадии клеточного цикла, когда в качестве неповреждённой гомологичной матрицы используются сестринские хроматиды[1]. В сравнении с гомологичными хромосомами, которые несут одни и те же гены, но разные аллели, сестринские хромосомы полностью идентичны друг другу и являются идеальными матрицами для рекомбинации. В свою очередь, НСК происходит во время G1-фазы клеточного цикла, когда клетка растёт, но хромосомы ещё не редуплицированы. Вне G1-фазы частота НСК намного меньше, однако его вероятность сохраняется на протяжении всего клеточного цикла. Механизмы, регулирующие и ГР, и НСК на протяжении всего цикла, варьируют в широких пределах у разных видов[26].
Циклинзависимые киназы (cdk), меняющие активность других белков при помощи фосфорилирования, играют важную роль в регуляции процесса ГР у эукариот[26]. У почкующихся дрожжей, когда начинается репликация ДНК, циклинзависимые киназы запускают ГР путём фосфорилирования белка Sae2[27]. Активированный таким образом Sae2 использует эндонуклеазы чтобы сделать точный двуцепочечный разрез в ДНК, после чего трёхкомпонентный гетеродимерный белок MRX связывается с ДНК и начинает белок-управляемую реакцию по обмену генетическим материалом между двумя молекулами ДНК[28].
Запуск пути НСК (NHEJ) начинается с привлечения к области повреждения белка 53BP1, который способствует дальнейшей репарации двуцепочечного разрыва по пути NHEJ. До момента подрезания концов возможно переключение на гомологичную рекомбинацию, которое достигается путём привлечения к повреждённой области белка-антагониста 53BP1 — BRCA1. Если BRCA1 вытесняет 53BP1, то двуцепочечный разрыв будет восстановлен по пути гомологичной рекомбинации[29]. Помимо 53BP1 и BRCA1, в выборе пути для устранения двуцепочечного разрыва задействованы белки RIF1 и CtIP — нуклеаза, задействованная в подрезании концов на первых этапах гомологичной рекомбинации. Таким образом, 53BP1 и RIF1 направляют восстановление по пути негомологичного соединения концов, а BRCA1 и CtIP — по пути гомологичной рекомбинации[30].
Гомологичная рекомбинация для восстановления двуцепочечных разрывов может быть использована только в S- и G2-фазах, когда в результате удвоения ДНК появляется матрица для репарации (поэтому NHEJ, активный во время всего клеточного цикла, является основным механизмом восстановления двуцепочечных разрывов в клетках млекопитающих). Исключение составляют области генома, содержащие повторы, например, повторы генов, кодирующих рРНК (рДНК). В рДНК матрица для восстановления двуцепочечного разрыва в повторе имеется в течение всего клеточного цикла, ею может выступать любой другой повтор. В случае рДНК мелкие повреждения быстро устраняются NHEJ внутри ядрышка (время протекания NHEJ составляет около 30 минут, а гомологичной рекомбинации — примерно 7 часов), а крупные и сложные повреждения перемещаются вместе с белками фибриллярных центров и плотного фибриллярного компонента на периферию, образуя так называемый ядрышковый кэп. В ядрышковом кэпе происходят все, кроме самых первых, этапы гомологичной рекомбинации, при этом повторы рДНК сближаются, что способствует рекомбинации. В ядрышковых кэпах NHEJ не происходит[31]. На выбор пути восстановления двуцепочечного разрыва также влияет сложность повреждения. NHEJ, как правило, используется для устранения небольших повреждений[32].
Модели[править | править код]
Известно два основных механизма гомологичной рекомбинации: путь репарации двуцепочечных разрывов (DSBR-путь), также известный как модель двойной структуры Холлидея, и путь синтезозависимого отжига цепи (SDSA-путь)[33]. Оба начинаются одинаковым образом. Когда двуцепочечный разрыв в цепи обнаружен, белковый комплекс MRX (у человека MRN) встает по обе стороны от разрыва, после чего следует отрезание 5'-концов, проходящее в два отдельных этапа. Первый этап заключается в том, что MRX в паре с белком Sae2 вырезают 5'-концы цепи около разрыва, тем самым оставляя выступающие 3'-концы. Второй этап 5' → 3'-отрезания продолжает геликаза Sgs1 и нуклеазы Exo1 и Dna2. Sgs1 «расстёгивает» двойную спираль, а Exo1 и Dna2 создают разрывы в одноцепочечной ДНК, высвобожденной Sgs1[27].
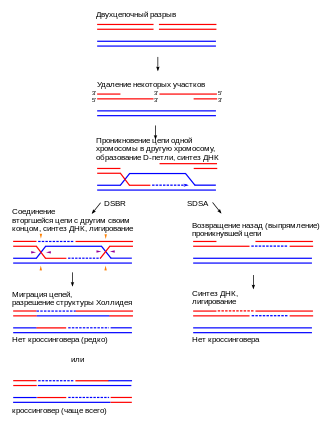
Репликативный белок А (RPA), имеющий высокое сродство к одноцепочечной ДНК, связывает выступающие 3'-концы[34] и с помощью ряда других белков, которые опосредуют процесс, например Rad51 (и Dmc1 в мейозе), формирует комплекс с одноцепочечной ДНК, покрывая её. Затем нуклеопротеидная нить ищет похожую или идентичную цепь ДНК и внедряется в неё, когда находит. В клетках, делящихся путём митоза, «жертвой» внедрения (реципиентным ДНК-дуплексом) обычно является сестринская хроматида, идентичная повреждённой ДНК, которая чаще всего используется в качестве матрицы для репарации. В мейозе, однако, реципиентным ДНК-дуплексом служит гомологичная хромосома, которая очень похожа на повреждённую хромосому, но не обязательно идентична ей[33].
В ходе вторжения цепи между торчащим 3'-концом внедряющейся цепи и гомологичной хромосомой образуется D-петля. После этого ДНК-полимераза продлевает 3'-концы. Получившаяся перекрёстная структура называется структурой Холлидея. Вслед за этим на внедрённой цепи (то есть на одном из выступающих 3'-концов) происходит синтез ДНК, эффективно восстанавливая её комплементарно гомологичной хромосоме в том месте, откуда была вытеснена D-петля[33].
Репарация двуцепочечных разрывов[править | править код]
После отрезания, внедрения нити в соседнюю хромосому и синтеза ДНК ДНК-полимеразой различия между путями репарации двуцепочечных разрывов (DSBR-путь) и синтез-зависимого отжига цепей (SDSA-путь) становятся более отчётливыми[33]. DSBR-путь уникален тем, что на втором выступающем 3'-конце (который не участвовал во внедрении) также образуется структура Холлидея с цепью гомологичной хромосомы. Далее двойная структура Холлидея становится продуктом рекомбинации под действием никирующих эндонуклеаз — рестриктаз, вносящих разрыв только в одну цепь ДНК. DSBR, как правило, влечёт за собой кроссинговер, хотя иногда конечный продукт может быть другим (не претерпевшим кроссинговер). С использованием плазмид и эндонуклеаз на примере митоза почкующихся дрожжей была показана способность повреждённой нуклеотидной цепи принимать последовательности нуклеотидов у других молекул ДНК[35][36]. Из-за тенденции к кроссинговеру DSBR-путь, вероятно, можно рассматривать как модель кроссинговера, происходящего во время мейоза[21].
Приведёт DSBR к кроссинговеру или нет, определяется тем, как будет разрезана, или «разрешена», структура Холлидея. Кроссинговер может произойти, если одна структура Холлидея будет разрезана по пересекающимся нитям, а другая нет. Продукт, не подвергшийся кроссинговеру, получится лишь в том случае, если обе структуры разрешены по пересекающимся нитям[1].
Синтез-зависимый отжиг цепей[править | править код]
Гомологичная рекомбинация путём SDSA (англ. synthesis-dependent strand annealing) приводит к образованию хромосом, не прошедших процесс кроссинговера. Сначала SDSA следует классическому сценарию: одна из цепей повреждённой ДНК внедряется в молекулу-матрицу, вытесняя из последней другую цепь, в результате чего образуется D-петля и структура Холлидея. Далее же ДНК-полимераза продлевает внедрённую цепь комплементарно молекуле-матрице, и одновременно с этим комплементарно вытесненной цепи синтезируется остальная часть повреждённой ДНК. При этом возможен процесс миграции точки ветвления, когда точка пересечения цепей, принадлежащих рекомбинирующим ДНК, начинает перемещаться между ними. Иногда в процессе слияния новосинтезированных цепей с молекулой ДНК могут возникать участки, выбивающиеся из дуплекса (двойной спирали), а также иные возможные пробелы и бреши. Все они успешно вырезаются и устраняются в процессе лигирования, после чего рекомбинацию можно считать завершённой[37].
Во время митоза именно SDSA-путь является основным ГР-путём репарации двуцепочечных разрывов ДНК[38] Тем не менее, при мейозе тоже часто происходит гомологичная рекомбинация без кроссинговера, которая, вероятно, является примером репарации разного рода повреждений[38][39].
Отжиг одиночной цепи[править | править код]

Путь отжига одиночной цепи (англ. single-strand annealing, SSA))[40] уникален тем, что, в отличие от DSBR и SDSA, он не требует наличия других молекул ДНК в процессе гомологичной рекомбинации. Поскольку участки цепи, для которых характерен SSA, состоят из повторяющихся последовательностей нуклеотидов, эти же последовательности и используются в качестве матриц, по которым строится недостающая часть цепи. SSA следует относительно простой схеме: после отрезания 5'-концов повреждённого участка цепи оставшиеся выступающие 3'-концы сближаются и сращиваются друг с другом, восстанавливая ДНК до прежнего вида[37][41].
По мере того как обрезаются участки повреждённой ДНК, образующиеся 3'-концы связываются с репликативным белком А, который предотвращает их спаривание друг с другом[42]. Затем белок Rad52 выравнивает обе цепи, чтобы дать комплементарным последовательностям образовать связи друг с другом[42]. Левозакрученные негомологичные участки ДНК, выбивающиеся из основного дуплекса, срезаются набором нуклеаз, известных как Rad1/Rad10, попадающих к ним с помощью белков Saw1 и Slx4[42][43]. Далее следует лигирование, которое заполняет любые оставшиеся пробелы в ДНК[44]. Процесс SSA считается мутагенным, так как в результате теряется какая-то часть ДНК, где происходила репарация[37].
Репликация, индуцированная разрывами[править | править код]
Двухцепочечные разрывы цепи иногда могут происходить во время репликации ДНК на так называемой репликационной вилке, образующейся, когда геликаза «расстёгивает» молекулу ДНК. Такие повреждения репарируются при помощи репликации, индуцированной разрывами (англ. break-induced replication, BIR) — ещё одного вида гомологичной рекомбинации, точные молекулярные механизмы которого всё ещё остаются неясными. На данный момент предложено три возможных варианта, и все они начинаются одинаково: одна из цепей повреждённой ДНК вторгается в соседнюю молекулу, однако механизм формирования D-петли и дальнейший ход событий у них различны[45].
Известно, что BIR-путь также может поддерживать длину теломер в отсутствие (или совместно с) теломеразы. Без рабочей теломеразы теломеры с каждым циклом митоза становятся короче, что в конечном итоге блокирует клеточный цикл и приводит к старению клетки. В почкующихся дрожжах, где теломераза была инактивирована мутациями, наблюдались два типа «выживших» клеток, которым удавалось избегать старения намного дольше, поддерживая длину теломер с помощью BIR[45].
Поддержание длины теломер крайне важно для обеспечения клеточного бессмертия, например, раковым клеткам. Клетки большинства видов рака избегают критического укорочения теломер с помощью высокого уровня экспрессии теломеразы. Тем не менее, есть виды рака, где опухолеобразование поддерживается альтернативными путями поддержания длины теломер[46]. Этот факт заставил учёных сконцентрироваться на вопросе о том, могут ли альтернативные механизмы, поддерживающие длину теломер, свести на нет эффект от некоторых противораковых препаратов, таких как ингибиторы теломеразы[47].
У прокариот[править | править код]


Хотя бактериальная гомологичная рекомбинация отличается от таковой у эукариот, она точно так же обеспечивает бактерий генетическим разнообразием и является для них основным механизмом репарации ДНК. Лучше всего процесс ГР изучен у E. coli[50]. Двуцепочечные и одноцепочечные повреждения бактериальной ДНК исправляются двумя разными путями: RecBCD и RecF соответственно[51]. Оба этих способа включают в себя серию реакций, известных как миграция точки ветвления, во время которой две двуцепочечные молекулы ДНК обмениваются одной из своих цепей, а также разрешение, когда эти две скрещивающиеся молекулы разрезаются на части и восстанавливаются до своего нормального двуцепочечного состояния.
RecBCD[править | править код]
RecBCD-путь — основной путь рекомбинации у бактерий, благодаря которому исправляются многие двуцепочечные разрывы, вызванные ультрафиолетовым и другого типа излучениями, а также различными химическими веществами[52][53][54]. Двуцепочечные повреждения нередко возникают в процессе репликации ДНК из одноцепочечных разрывов, что приводит к коллапсу репликативной вилки, и репарируются несколькими ГР-путями, включая RecBCD[55].
Фермент RecBCD, состоящий из трёх субъединиц, инициирует рекомбинацию путём связывания с тупым или почти тупым концом ДНК-дуплекса (тупым называется конец, где ни одна из двух цепей не выступает за пределы молекулы). Дальше субъединицы RecB и RecD, имеющие геликазную активность, расплетают дуплекс, а RecB при этом может функционировать ещё и как нуклеаза. Дуплекс расплетается вплоть до того момента, когда RecBCD сталкивается с определённой нуклеотидной последовательностью (5'-GCTGGTGG-3'), известной как Chi-сайт[54].
Столкновение с Chi-сайтом резко меняет активность фермента RecBCD[53][48][56]. Раскручивание цепи замирает на несколько секунд, а затем возобновляется со скоростью, примерно вполовину меньшей, чем вначале. Вероятно, это связано с тем, что после Chi-сайта ДНК расплетает хеликаза RecB — более медленная, чем RecD, которая расплетает ДНК до Chi-сайта[57][58]. Распознавание Chi-сайта приводит к тому, что RecBCD вносит разрыв в цепь, содержащую Chi-сайт, и начинает загружать белки RecA на новообразованный 3'-конец. Получившаяся нуклеопротеидная нить ищет похожую последовательность ДНК на гомологичной хромосоме и внедряется в неё. Процесс поиска вызывает растяжение ДНК-дуплекса, что усиливает гомологическую распознаваемость (механизм, называемый конформационным считыванием (англ. Conformational proofreading))[59][60][61]. Внедрение нуклеопротеидной нити вытесняет одну из цепей дуплекса гомологичной хромосомы и формируется D-петля, дальнейший разрез которой приведёт к появлению структуры Холлидея[54]. Если две взаимодействующие молекулы различаются, то разрешение структуры при помощи белков RuvABC или RecG создаёт две рекомбинантные молекулы ДНК разных генетических типов (реципрокный механизм). Однако возможен и альтернативный ход развития событий: внедрение 3'-нуклеопротеидной цепи с Chi-сайтом на конце может спровоцировать синтез ДНК и образование репликативнной вилки, но в итоге образуется только один тип рекомбинантной ДНК (нереципрокный механизм)[48].
RecF[править | править код]
Путь RecF используется бактериями для репарации одноцепочечных повреждений, однако, в том случае когда мутации инактивируют белок RecBCD и нуклеазы SbcCD и ExoI, этим путём могут восстанавливаться и двойные разрывы в ДНК[62]. В ходе RecF хеликаза RecQ раскручивает ДНК, а нуклеаза RecJ разрушает цепь, обращённую 5'-концом к ферменту, оставляя цепь, обращённую 3'-концом, нетронутой. Далее на эту цепь садится множество белков RecA при содействии таких белков, как RecF, RecO и RecR. Получившаяся нуклеопротеидная нить ищет гомологичную ДНК-матрицу и обменивается с ней идентичной или приблизительно идентичной последовательностью нуклеотидов[63].
Хотя белки и специфические механизмы, участвующие в путях RecBCD и RecF, различны, оба пути основаны на внедрении 3'-направленного нуклеопротеида и оба включают в себя такие процессы, как миграция точки ветвления, образование структуры Холлидея и разрешение этой структуры как реципрокного, так и нереципрокного типа[64][63].
Миграция точки ветвления[править | править код]
Сразу после внедрения цепи образовавшаяся структура Холлидея начинает движение вдоль ДНК-дуплексов, между которыми в этот момент происходит обмен парами оснований. Для катализа миграции ветвления белок RuvA распознаёт и связывается со структурой Холлидея, после чего привлекает к процессу белок RuvB, формируя RuvAB-комплекс. Два набора RuvB белка, каждый из которых образует кольцевые ATPазы, загружаются на противоположные стороны структуры Холлидея, где они выступают как два насоса, закачивающие энергию, требующуюся в процессе миграции ветвления. Далее два комплекта белка RuvA собираются между кольцами RuvB в центре структуры Холлидея таким образом, что ДНК в структуре оказывается зажатой между ними. Два рекомбинирующих дуплекса расплетаются под действием RuvA и обмениваются нуклеотидными последовательностями[65][66].
Разрешение[править | править код]
На стадии разрешения рекомбинации все структуры Холлидея, сформированные во время внедрения нити, расщепляются определённым образом, разделяя две молекулы ДНК. Это расщепление осуществляется RuvAB, взаимодействующим с RuvC, которые в совокупности образуют RuvABC-комплекс. RuvC — это эндонуклеаза, которая вырезает вырожденную последовательность нуклеотидов 5'-(A/Т)ТТ(G/C)-3', которая встречается в ДНК примерно раз в 64 нуклеотида[66]. Прежде чем резать, RuvC, вероятно, получает доступ к структуре Холлидея, вытесняя один из двух тетрамеров RuvA, покрывая в этом месте ДНК[65]. В результате рекомбинации образуется либо сплайс-продукт, либо патч-продукт, в зависимости от того, каким образом структура Холлидея была разрезана RuvC-белком[66]. Сплайс-продуктом называется тот, который прошёл процесс кроссинговера, в котором произошла перестройка генетического материала вокруг всего сайта рекомбинации. Патч-продукты кроссинговеру не подвергаются и перестраивается лишь незначительная часть цепи[67].
Содействие переносу генов[править | править код]
Гомологичная рекомбинация — важный метод по интегрированию ДНК донора в геном реципиента во время горизонтального переноса генов. Обычно рекомбинация при горизонтальной передаче генов происходит только между похожими бактериями, поскольку для неё требуется, чтобы ДНК донора и реципиента были очень похожи[68]. Исследования, проведённые на нескольких видах бактерий, установили, что существует полулогарифмическая зависимость между разницей в последовательностях ДНК донора и реципиента и частотой рекомбинаций. Последняя тем ниже, чем выше различие в геноме донора и реципиента[69][70][71].
В бактериальной конъюгации, где ДНК передаётся между бактериями посредством прямого межклеточного контакта, гомологичная рекомбинация способствует интеграции чужеродной ДНК в геном через RecBCD-путь. Фермент RecBCD способствует рекомбинации, после того как ДНК преобразуется из одноцепочечной формы, какой она изначально попала в бактерию, в двухцепочечную во время репликации. RecBCD также необходим для заключительного этапа трансдукции, когда горизонтальный перенос генов между бактериями осуществляется с помощью вируса-бактериофага. Бактериальная ДНК переносится вирусом в капсидной головке, куда она иногда может неправильно упаковываться так, как упаковывается вирусная ДНК во время репликации фага. Когда вирус инфицирует другую бактерию, ДНК прежней бактерии-хозяина попадает в клетку уже в форме двойной спирали, где включается ферментом RevBCD в геном нового хозяина[54].
Бактериальная трансформация[править | править код]
Естественная бактериальная трансформация предполагает передачу ДНК от бактерии донора к бактерии реципиенту, где и донор, и реципиент, как правило, одного вида. Трансформация, в отличие от бактериальной конъюгации и трансдукции, зависит от многих бактериальных генных продуктов, которые специфически взаимодействуют в ходе процесса[72]. Таким образом, трансформация — это явно бактериальный механизм адаптации для передачи ДНК. Для того чтобы бактерия могла взять и интегрировать ДНК донора в хромосому путём гомологичной рекомбинации, она должна сначала войти в особое физиологическое состояние, называемое компетенцией. Семейство-белков RecA/Rad51/DMC1 играет центральную роль при гомологичной рекомбинации во время трансформации, как это происходит в эукариотических мейозе и митозе. Например, белок RecA необходим для трансформации у таких бактерий, как Bacillus subtilis и Streptococcus pneumoniae[73].
Как часть процесса трансформации белок RecA взаимодействует с вводящейся одноцепочечной ДНК (оцДНК) в форме RecA/оцДНК-нуклеофиламента, который сканирует местную хромосому с целью выявления гомологичных областей и доводит к ним оцДНК, где и происходит гомологичная рекомбинация[74].
У вирусов[править | править код]
Гомологичная рекомбинация характерна для нескольких групп вирусов. В ДНК таких вирусов, как вирус герпеса, рекомбинация происходит тем же образом, что у эукариот и бактерий[75]. Известно, что РНК-содержащие вирусы могут иметь геном положительной полярности или отрицательной полярности. Существуют данные о рекомбинации у вирусов, чей геном представлен одноцепочечной РНК положительной полярности, таких как ретровирусы, пикорнавирусы и коронавирусы, однако неизвестно, происходит ли гомологичная рекомбинация в РНК-вирусах с геномом отрицательной полярности, например, у вируса гриппа[76].
Рекомбинация в РНК-содержащих вирусах может быть точной и неточной. В первом случае, в РНК-РНК-рекомбинации, нет разницы между двумя родительскими РНК-последовательностями, как и в вытекающим из процесса рекомбинации кроссинговере. Из-за этого часто трудно определить расположение последовательностей, прошедших кроссинговер. В неточной рекомбинации кроссинговер определить намного проще, поскольку можно проследить добавление новых нуклеотидов, делецию и иные модификации. Уровень точности процесса зависит от последовательности рекомбинирующих молекул РНК: последовательность, богатая аденином и урацилом, снижает точность кроссинговера[77][78].
Гомологичная рекомбинация важна для эволюции вирусов[77][79]. Например, если геномы двух вирусов с различными невыгодными мутациями подвергаются рекомбинации, то они способны образовать другой, полностью функциональный геном, а в случае, когда два похожих вируса заражают одну и ту же клетку, их гомологичная рекомбинация может привести к удачному обмену генов и тем самым создать более мощные варианты себя самих[79].
Кроме того, гомологичная рекомбинация предлагается как механизм, посредством которого ДНК-содержащий вирус герпеса человека-6 интегрируется в человеческие теломеры[80].
Когда два или более вирусов, каждый из которых содержит смертельные для него геномные повреждения, заражают одну клетку-хозяина, вирусные геномы часто проходят спаривание друг с другом и подвергаются репарации, создавая тем самым жизнеспособный дочерний фаг. Этот процесс, известный как кратность реактивации, был изучен у нескольких бактериофагов, в том числе у фага Т4[81]. Ферменты, вовлечённые в процесс репарации у фага T4, функционально гомологичны ферментам бактерий и эукариот[82]. В отношении гена, необходимого для реакции обмена нитей, ключевого шага в гомологичной рекомбинативной репарации, прослеживается функциональная гомология от вирусов до человека (uvsX у фага Т4; RecA в E. coli и других бактериях, и rad51 и dmc1 у дрожжей и других эукариот, включая человека)[83]. Кратность реактивации также была продемонстрирована в многочисленных патогенных вирусах[84].
Последствия дисфункции[править | править код]
Без надлежащей гомологичной рекомбинации хромосомы часто неправильно выстраиваются в первой фазе клеточного деления мейоза, из-за чего происходит нерасхождение и хромосомы неправильно разделяются. В свою очередь, нерасхождение может привести к тому, что сперматозоид или яйцеклетка будут иметь слишком мало или слишком много хромосом. Синдром Дауна, который вызывается дополнительной копией 21-хромосомы — лишь одно из многих нарушений, которые возникают в результате такого сбоя процесса гомологичной рекомбинации в мейозе[66][85].
Канцерогенез у людей часто бывает следствием дефектов в механизме гомологичной рекомбинации. Например, такие заболевания как синдром Блума, синдром Вернера и синдром Ротмунда — Томпсона, вызваны неисправностями генов, кодирующих участвующие в регуляции процесса ГР белки: BLM, WRN и RECQ4 соответственно[86]. В клетках пациентов с синдромом Блума, у которых нет рабочей копии белка BLM, скорость гомологичной рекомбинации повышена в сравнении с нормой[87]. Опыты на мышах, дефицитных по BLM, заставили предположить, что эта мутация вызывает рак через потерю гетерозиготности, вызванную повышенным уровнем гомологичной рекомбинации[88]. Потеря гетерозиготности — это потеря одного из аллелей определённого гена. Если утраченный аллель способствует подавлению опухоли, как, к примеру ген белка ретинобластомы, то такая потеря гетерозиготности может привести к раку[1].
Эффективность репарации ДНК падает со снижением скорости гомологической рекомбинации[1], что также может привести к раку[89], например, в случае BRCA1 и BRCA2 — двух похожих опухолевых супрессоров, чья неисправность связана с значительно повышенной вероятностью возникновения рака груди и яичников. Клетки с такой неисправностью имеют пониженный уровень гомологичной рекомбинации и бо́льшую чувствительность к ионизирующему излучению, что неизбежно означает повышенную восприимчивость к раку[89]. Поскольку единственная известная функция BRCA2 — облегчение инициации гомологичной рекомбинации, исследователи предположили, что более детальное исследование этого белка может быть ключом к пониманию причин рака молочной железы и яичников[89].
Эволюционная консервативность[править | править код]

Хотя механизм осуществления рекомбинации сильно варьируется, он имеется во всех доменах жизни[90]. На основе сходства их аминокислотных последовательностей, гомологи ряда белков могут быть обнаружены в различных доменах жизни, показывая тем самым, что они появились очень давно и с тех пор эволюционировали от общих предков белков[90].
Семейство белков рекомбиназы RecA обнаруживается почти во всех организмах: RecA у бактерий, Rad51 и DMC1 у эукариот, RadA у архей и UvsX у фага T4[91]. Во всех трёх доменах прослеживаются родственные белки, связывающие одноцепочечную ДНК, которые играют роль в рекомбинации и многих других процессах[92]; Rad54, Mre11, Rad50 и ряд других белков также найдены у архей и эукариот[90][91][93].
Семейство белков рекомбиназы RecA[править | править код]
Белки семейства RecA, как считается, произошли от общего рекомбиназного предка. В эту семью входят RecA-белки бактерий, Rad51- и Dmc1-белки эукариот, и RadA-белки архей и ряд белков-паралогов. Исследования моделирования эволюционных связей между Rad51, Dmc1 и RadA свидетельствуют о том, что они имеют общего молекулярного предка. В рамках этого белкового семейства Rad51 и Dmc1 группируются в отдельную от RadA кладу. Одна из причин для группирования этих трёх белков состоит в том, что все они обладают модифицированным мотивом спираль-поворот-спираль, который помогает белкам связываться с ДНК по направлению к их N-концу[90]. Древняя дупликация эукариотического RecA и последующие мутации были предложены в качестве вероятного происхождения современных генов Rad51 и Dmc1[90].
Эти белки обычно имеют длинные консервативные последовательности, известные как RecA/Rad51-домен, который содержит две последовательности мотивов: Уолкер-А мотив и Уолкер-Б мотив. А- и Б-мотивы дают возможность членам домена RecA/Rad51 связывать и гидролизовать АТФ[90][94].
Мейоз-специфические белки[править | править код]
Открытие белка Dmc1 в нескольких видах Лямблий, одних из первых простейших эукариот, говорит о том, что мейотическая гомологичная рекомбинация и, таким образом, мейоз сам по себе, возникли очень рано в эволюции эукариот[95]. В дополнение к исследованиям Dmc1, исследования белка Spo11 предоставили информацию о происхождении мейотической рекомбинации[96]. Spo11 (топоизомераза II типа) может инициировать гомологичную рекомбинацию в процессе мейоза посредством создания нацеленных двуцепочечных разрывов в ДНК[23]. Филогенетические деревья, основанные на последовательности генов Spo11, похожи у животных, грибов, растений, протистов и архей, и привели учёных к убеждению, что современная версия Spo11 появились у последнего общего предка эукариот и архей[96].
Применение в технологии[править | править код]
Генетический таргетинг[править | править код]

Множество методов по введению последовательностей ДНК в организм для создания рекомбинантных ДНК и генетически модифицированных организмов используют процесс гомологичной рекомбинации[97]. Также называемый генетическим таргетингом, метод особенно распространен в генетике дрожжей и мышей. Метод генетического таргетинга в нокаутных (генетически модифицированных) мышах посредством эмбриональных стволовых клеток поставляет генетический материал (в основном в терапевтических интересах), который подавляет целевой ген мыши по принципу гомологичной рекомбинации. Мышь таким образом выступает в качестве рабочей модели, по которой можно понять работу конкретных генов млекопитающих. Марио Капекки, Мартин Эванс и Оливер Смитис были удостоены в 2007 году Нобелевской премии по физиологии и медицине за то, что они выяснили, как можно использовать гомологичную рекомбинацию чтобы редактировать мышиный геном[98].
Достижения в технологиях генетического таргетинга, использующего механизм гомологичной рекомбинации, привели к развитию новой волны более точных изогенных моделей человеческих заболеваний (то есть клеток, отбирающихся или проектирующихся для создания более точной генетической модели наследственных заболеваний). Эти спроектированные человеческие клетки-модели более точно отражают генетику болезней, чем их мышиные предшественники, что обусловлено, по большей части, интересом к эндогенным мутациям, происходящим так же, как это происходит у реальных пациентов, а также тем фактом, что они основаны на человеческом геноме, а не на мышином. Кроме того, некоторые технологии позволяют использовать метод нок-ин в конкретных мутациях, а не только нокаут, как это было в старых версиях генетического таргетинга[99].
Белковая инженерия[править | править код]
Белковая инженерия с гомологичной рекомбинацией создаёт химерные белки путём обмена фрагментами двух родительских белков. Эти методы используют тот факт, что рекомбинация может привести к высокой степени многообразия последовательностей при сохранении способности белков к фолдингу[100]. Это создаёт контраст с другими методами белковой инженерии, такими как случайный точечный мутагенез, в которых вероятность сохранения функции белков снижается в геометрической прогрессии с увеличением количества аминокислотных замен[101]. Создаваемые химеры сохраняют способность к нормальному функционированию благодаря тому, что родительские фрагменты имеют высокую структурную и эволюционную консервативность. Эти рекомбинантные строительные блоки сохраняют структурно-важные взаимодействия, вроде точек физического контакта аминокислот. Вычислительные методы, такие как SCHEMA и статистический анализ связи (SCA) могут быть использованы для определения структурных фрагментов, подходящих для рекомбинации[102][103][104].
Методы, основанные на гомологичной рекомбинации, используются для создания новых белков[102]. В исследовании, опубликованном в 2007 году, исследователям удалось создавать химеру из двух ферментов, участвующих в биосинтезе изопреноидов — разнообразного класса соединений, включая гормоны, зрительные пигменты и определённые феромоны. Химерные белки приобрели способность катализировать важные реакции изопреноидного биосинтеза — одного из самых разнообразных путей биосинтеза в природе, то есть способность, отсутствовавшую в родительских белках[105]. Белковая инженерия на основе рекомбинации также создаёт химерные ферменты с новыми функциями, члены группы белков, известной как семейство цитохром Р450[106], который в организме человека принимает участие в детоксикации чужеродных соединений типа наркотиков, лекарств, пищевых добавок и консервантов[21].
Терапия рака[править | править код]

Раковые клетки с BRCA-мутациями имеют нарушения в процессе в гомологичной рекомбинации, и препараты, использующие эти недостатки, успешно разрабатываются и применяются для терапии раковых заболеваний[107][108]. Олапариб, ингибитор PARP1, подавляет или полностью прекращает рост опухолей в случае рака молочной железы, рака яичника и рака предстательной железы, которые были вызваны мутациями в генах BRCA1 или BRCA2, необходимых для ГР. Если BRCA1 или BRCA2 отсутствует, другие типы репарации ДНК должны компенсировать этот недостаток, например эксцизионная репарация оснований (BER) для починки повреждений репликативной вилки или негомологичное соединение концов в случае двухцепочечных разрывов[107]. Путём ингибирования BER в ГР-дефицитных клетках олапариб задействует принцип синтетической летальности (комбинация двух или более мутаций, приводящих к смерти клетки) для уничтожения раковых клеток. Хоть ингибиторы PARP1 представляют собой новый подход к терапии рака, учёные говорят, что они могут оказаться неэффективными в лечении поздних стадий метастатического рака[107]. Раковые клетки могут стать устойчивыми к ингибиторам PARP1, если они подвергаются делеции во время мутации гена BRCA2, таким образом восстанавливая способность к гомологичной рекомбинации и подрывая эффект синтетической летальности[109].
Примечания[править | править код]
- ↑ 1 2 3 4 5 6 7 Альбертс и др., 2013, с. 466—484.
- ↑ Capecchi M. R. Altering the genome by homologous recombination. (англ.) // Science (New York, N.Y.). — 1989. — Vol. 244, no. 4910. — P. 1288—1292. — PMID 2660260.
- ↑ Smithies O., Gregg R. G., Boggs S. S., Koralewski M. A., Kucherlapati R. S. Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination. (англ.) // Nature. — 1985. — Vol. 317, no. 6034. — P. 230—234. — PMID 2995814.
- ↑ Orr-Weaver T. L., Szostak J. W., Rothstein R. J. Yeast transformation: a model system for the study of recombination. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 1981. — Vol. 78, no. 10. — P. 6354—6358. — PMID 6273866.
- ↑ Orr-Weaver T. L., Szostak J. W. Yeast recombination: the association between double-strand gap repair and crossing-over. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 1983. — Vol. 80, no. 14. — P. 4417—4421. — PMID 6308623.
- ↑ 1 2 Szostak J. W., Orr-Weaver T. L., Rothstein R. J., Stahl F. W. The double-strand-break repair model for recombination. (англ.) // Cell. — 1983. — Vol. 33, no. 1. — P. 25—35. — PMID 6380756.
- ↑ Resnick M. A. The repair of double-strand breaks in DNA; a model involving recombination. (англ.) // Journal of theoretical biology. — 1976. — Vol. 59, no. 1. — P. 97—106. — PMID 940351.
- ↑ Jasin M., Rothstein R. Repair of strand breaks by homologous recombination. (англ.) // Cold Spring Harbor perspectives in biology. — 2013. — Vol. 5, no. 11. — P. 012740. — doi:10.1101/cshperspect.a012740. — PMID 24097900.
- ↑ Bateson P. William Bateson: a biologist ahead of his time. (англ.) // Journal of genetics. — 2002. — Vol. 81, no. 2. — P. 49—58. — PMID 12532036.
- ↑ Reginald Crundall Punnett. NAHSTE, University of Edinburgh. Дата обращения: 3 июля 2010. Архивировано 25 ноября 2010 года.
- ↑ Lobo I., Shaw K. Thomas Hunt Morgan, genetic recombination, and gene mapping (англ.) // Nature Education : journal. — 2008. — Vol. 1, no. 1. Архивировано 22 августа 2016 года.
- ↑ 1 2 Coe E., Kass L. B. Proof of physical exchange of genes on the chromosomes. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2005. — Vol. 102, no. 19. — P. 6641—6646. — doi:10.1073/pnas.0407340102. — PMID 15867161.
- ↑ Creighton H. B., McClintock B. A Correlation of Cytological and Genetical Crossing-Over in Zea Mays. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 1931. — Vol. 17, no. 8. — P. 492—497. — PMID 16587654.
- ↑ Stern C. Zytologisch-genetische untersuchungen alsbeweise fur die Morgansche theorie des faktoraustauschs (нем.) // Biol. Zentbl. : magazin. — 1931. — Bd. 51. — S. 547—587.
- ↑ The development of bacterial genetics. US National Library of Medicine. Дата обращения: 3 июля 2010. Архивировано 9 июня 2010 года.
- ↑ The Nobel Prize in Physiology or Medicine 1958. Nobelprize.org. Дата обращения: 3 июля 2010. Архивировано 19 февраля 2007 года.
- ↑ 1 2 3 Haber J. E., Ira G., Malkova A., Sugawara N. Repairing a double-strand chromosome break by homologous recombination: revisiting Robin Holliday's model. (англ.) // Philosophical transactions of the Royal Society of London. Series B, Biological sciences. — 2004. — Vol. 359, no. 1441. — P. 79—86. — doi:10.1098/rstb.2003.1367. — PMID 15065659.
- ↑ Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. 12.5: Recombination between Homologous DNA Sites: Double-Strand Breaks in DNA Initiate Recombination // Molecular Cell Biology (неопр.). — 4th. — W. H. Freeman and Company, 2000. — ISBN 0-7167-3136-3.
- ↑ Griffiths AJF et al. 8: Chromosome Mutations: Chromosomal Rearrangements // Modern Genetic Analysis (неопр.). — W. H. Freeman and Company, 1999. — ISBN 0-7167-3118-5. Архивировано 27 февраля 2009 года.
- ↑ Khanna K. K., Jackson S. P. DNA double-strand breaks: signaling, repair and the cancer connection. (англ.) // Nature genetics. — 2001. — Vol. 27, no. 3. — P. 247—254. — doi:10.1038/85798. — PMID 11242102.
- ↑ 1 2 3 Nelson D. L., Cox MM. Principles of Biochemistry (неопр.). — 4th. — Freeman, 2005. — С. 980—981. — ISBN 978-0-7167-4339-2.
- ↑ Marcon E., Moens P. B. The evolution of meiosis: recruitment and modification of somatic DNA-repair proteins. (англ.) // BioEssays : news and reviews in molecular, cellular and developmental biology. — 2005. — Vol. 27, no. 8. — P. 795—808. — doi:10.1002/bies.20264. — PMID 16015600.
- ↑ 1 2 Keeney S., Giroux C. N., Kleckner N. Meiosis-specific DNA double-strand breaks are catalyzed by Spo11, a member of a widely conserved protein family. (англ.) // Cell. — 1997. — Vol. 88, no. 3. — P. 375—384. — PMID 9039264.
- ↑ Longhese M. P., Bonetti D., Guerini I., Manfrini N., Clerici M. DNA double-strand breaks in meiosis: checking their formation, processing and repair. (англ.) // DNA repair. — 2009. — Vol. 8, no. 9. — P. 1127—1138. — doi:10.1016/j.dnarep.2009.04.005. — PMID 19464965.
- ↑ Cahill L. P., Mariana J. C., Mauléon P. Total follicular populations in ewes of high and low ovulation rates. (англ.) // Journal of reproduction and fertility. — 1979. — Vol. 55, no. 1. — P. 27—36. — PMID 423159.
- ↑ 1 2 Shrivastav M., De Haro L. P., Nickoloff J. A. Regulation of DNA double-strand break repair pathway choice. (англ.) // Cell research. — 2008. — Vol. 18, no. 1. — P. 134—147. — doi:10.1038/cr.2007.111. — PMID 18157161.
- ↑ 1 2 Mimitou E. P., Symington L. S. Nucleases and helicases take center stage in homologous recombination. (англ.) // Trends in biochemical sciences. — 2009. — Vol. 34, no. 5. — P. 264—272. — doi:10.1016/j.tibs.2009.01.010. — PMID 19375328.
- ↑ Huertas P., Cortés-Ledesma F., Sartori A. A., Aguilera A., Jackson S. P. CDK targets Sae2 to control DNA-end resection and homologous recombination. (англ.) // Nature. — 2008. — Vol. 455, no. 7213. — P. 689—692. — doi:10.1038/nature07215. — PMID 18716619.
- ↑ Ragu Sandrine, Matos-Rodrigues Gabriel, Thomas Melissa, Lopez Bernard S. Homologous recombination in mammalian cells: From molecular mechanisms to pathology (англ.) // Genome Stability. — 2021. — P. 367—392. — doi:10.1016/B978-0-323-85679-9.00020-9.
- ↑ Decottignies A. Alternative end-joining mechanisms: a historical perspective. (англ.) // Frontiers In Genetics. — 2013. — Vol. 4. — P. 48—48. — doi:10.3389/fgene.2013.00048. — PMID 23565119.
- ↑ Blokhina Yana P., Buchwalter Abigail. Moving fast and breaking things: Incidence and repair of DNA damage within ribosomal DNA repeats (англ.) // Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. — 2020. — May (vol. 821). — P. 111715. — ISSN 0027-5107. — doi:10.1016/j.mrfmmm.2020.111715.
- ↑ Shibata A., Conrad S., Birraux J., Geuting V., Barton O., Ismail A., Kakarougkas A., Meek K., Taucher-Scholz G., Löbrich M., Jeggo P. A. Factors determining DNA double-strand break repair pathway choice in G2 phase. (англ.) // The EMBO Journal. — 2011. — 16 March (vol. 30, no. 6). — P. 1079—1092. — doi:10.1038/emboj.2011.27. — PMID 21317870.
- ↑ 1 2 3 4 Sung P., Klein H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. (англ.) // Nature reviews. Molecular cell biology. — 2006. — Vol. 7, no. 10. — P. 739—750. — doi:10. 1038/nrm2008. — PMID 16926856.
- ↑ Wold M. S. Replication protein A: a heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. (англ.) // Annual review of biochemistry. — 1997. — Vol. 66. — P. 61—92. — doi:10.1146/annurev.biochem.66.1.61. — PMID 9242902.
- ↑ McMahill M. S., Sham C. W., Bishop D. K. Synthesis-dependent strand annealing in meiosis. (англ.) // Public Library of Science Biology. — 2007. — Vol. 5, no. 11. — P. e299. — doi:10.1371/journal.pbio.0050299. — PMID 17988174.
- ↑ Bärtsch S., Kang L. E., Symington L. S. RAD51 is required for the repair of plasmid double-stranded DNA gaps from either plasmid or chromosomal templates. (англ.) // Molecular and cellular biology. — 2000. — Vol. 20, no. 4. — P. 1194—1205. — PMID 10648605.
- ↑ 1 2 3 Helleday T., Lo J., van Gent D. C., Engelward B. P. DNA double-strand break repair: from mechanistic understanding to cancer treatment. (англ.) // DNA repair. — 2007. — Vol. 6, no. 7. — P. 923—935. — doi:10.1016/j.dnarep.2007.02.006. — PMID 17363343.
- ↑ 1 2 Andersen S. L., Sekelsky J. Meiotic versus mitotic recombination: two different routes for double-strand break repair: the different functions of meiotic versus mitotic DSB repair are reflected in different pathway usage and different outcomes. (англ.) // BioEssays : news and reviews in molecular, cellular and developmental biology. — 2010. — Vol. 32, no. 12. — P. 1058—1066. — doi:10.1002/bies.201000087. — PMID 20967781.
- ↑ Allers T., Lichten M. Differential timing and control of noncrossover and crossover recombination during meiosis. (англ.) // Cell. — 2001. — Vol. 106, no. 1. — P. 47—57. — PMID 11461701.
- ↑ Разин С. В., Быстрицкий А. А. Хроматин: упакованный геном. — М.: БИНОМ. Лаборатория знаний, 2013. — С. 148. — 172 с. — ISBN 978-5-9963-1611-3.
- ↑ Haber lab. Single-strand annealing. "Brandeis University". Дата обращения: 3 июля 2010. Архивировано 19 января 2015 года.
- ↑ 1 2 3 Lyndaker A. M., Alani E. A tale of tails: insights into the coordination of 3' end processing during homologous recombination. (англ.) // BioEssays : news and reviews in molecular, cellular and developmental biology. — 2009. — Vol. 31, no. 3. — P. 315—321. — doi:10.1002/bies.200800195. — PMID 19260026.
- ↑ Mimitou E. P., Symington L. S. DNA end resection: many nucleases make light work. (англ.) // DNA repair. — 2009. — Vol. 8, no. 9. — P. 983—995. — doi:10.1016/j.dnarep.2009.04.017. — PMID 19473888.
- ↑ Pâques F., Haber J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. (англ.) // Microbiology and molecular biology reviews : MMBR. — 1999. — Vol. 63, no. 2. — P. 349—404. — PMID 10357855.
- ↑ 1 2 McEachern M. J., Haber J. E. Break-induced replication and recombinational telomere elongation in yeast. (англ.) // Annual review of biochemistry. — 2006. — Vol. 75. — P. 111—135. — doi:10.1146/annurev.biochem.74.082803.133234. — PMID 16756487.
- ↑ Morrish T. A., Greider C. W. Short telomeres initiate telomere recombination in primary and tumor cells. (англ.) // PLoS genetics. — 2009. — Vol. 5, no. 1. — P. e1000357. — doi:10.1371/journal.pgen.1000357. — PMID 19180191.
- ↑ Muntoni A., Reddel R. R. The first molecular details of ALT in human tumor cells. (англ.) // Human molecular genetics. — 2005. — Vol. 14 Spec No. 2. — P. 191—196. — doi:10.1093/hmg/ddi266. — PMID 16244317.
- ↑ 1 2 3 Amundsen S. K., Taylor A. F., Reddy M., Smith G. R. Intersubunit signaling in RecBCD enzyme, a complex protein machine regulated by Chi hot spots. (англ.) // Genes & development. — 2007. — Vol. 21, no. 24. — P. 3296—3307. — doi:10.1101/gad.1605807. — PMID 18079176.
- ↑ Singleton M. R., Dillingham M. S., Gaudier M., Kowalczykowski S. C., Wigley D. B. Crystal structure of RecBCD enzyme reveals a machine for processing DNA breaks. (англ.) // Nature. — 2004. — Vol. 432, no. 7014. — P. 187—193. — doi:10.1038/nature02988. — PMID 15538360.
- ↑ Kowalczykowski S. C., Dixon D. A., Eggleston A. K., Lauder S. D., Rehrauer W. M. Biochemistry of homologous recombination in Escherichia coli. (англ.) // Microbiological reviews. — 1994. — Vol. 58, no. 3. — P. 401—465. — PMID 7968921.
- ↑ Rocha E. P., Cornet E., Michel B. Comparative and evolutionary analysis of the bacterial homologous recombination systems. (англ.) // PLoS genetics. — 2005. — Vol. 1, no. 2. — P. e15. — doi:10.1371/journal.pgen.0010015. — PMID 16132081.
- ↑ Cromie G. A. Phylogenetic ubiquity and shuffling of the bacterial RecBCD and AddAB recombination complexes. (англ.) // Journal of bacteriology. — 2009. — Vol. 191, no. 16. — P. 5076—5084. — doi:10.1128/JB.00254-09. — PMID 19542287.
- ↑ 1 2 Smith G. R. How RecBCD enzyme and Chi promote DNA break repair and recombination: a molecular biologist's view. (англ.) // Microbiology and molecular biology reviews : MMBR. — 2012. — Vol. 76, no. 2. — P. 217—228. — doi:10.1128/MMBR.05026-11. — PMID 22688812.
- ↑ 1 2 3 4 Dillingham M. S., Kowalczykowski S. C. RecBCD enzyme and the repair of double-stranded DNA breaks. (англ.) // Microbiology and molecular biology reviews : MMBR. — 2008. — Vol. 72, no. 4. — P. 642—671. — doi:10.1128/MMBR.00020-08. — PMID 19052323.
- ↑ Michel B., Boubakri H., Baharoglu Z., LeMasson M., Lestini R. Recombination proteins and rescue of arrested replication forks. (англ.) // DNA repair. — 2007. — Vol. 6, no. 7. — P. 967—980. — doi:10.1016/j.dnarep.2007.02.016. — PMID 17395553.
- ↑ Spies M., Bianco P. R., Dillingham M. S., Handa N., Baskin R. J., Kowalczykowski S. C. A molecular throttle: the recombination hotspot chi controls DNA translocation by the RecBCD helicase. (англ.) // Cell. — 2003. — Vol. 114, no. 5. — P. 647—654. — PMID 13678587.
- ↑ Taylor A. F., Smith G. R. RecBCD enzyme is a DNA helicase with fast and slow motors of opposite polarity. (англ.) // Nature. — 2003. — Vol. 423, no. 6942. — P. 889—893. — doi:10.1038/nature01674. — PMID 12815437.
- ↑ Spies M., Amitani I., Baskin R. J., Kowalczykowski S. C. RecBCD enzyme switches lead motor subunits in response to chi recognition. (англ.) // Cell. — 2007. — Vol. 131, no. 4. — P. 694—705. — doi:10.1016/j.cell.2007.09.023. — PMID 18022364.
- ↑ Savir Y., Tlusty T. RecA-mediated homology search as a nearly optimal signal detection system. (англ.) // Molecular cell. — 2010. — Vol. 40, no. 3. — P. 388—396. — doi:10.1016/j.molcel.2010.10.020. — PMID 21070965.
- ↑ Rambo R. P., Williams G. J., Tainer J. A. Achieving fidelity in homologous recombination despite extreme complexity: informed decisions by molecular profiling. (англ.) // Molecular cell. — 2010. — Vol. 40, no. 3. — P. 347—348. — doi:10.1016/j.molcel.2010.10.032. — PMID 21070960.
- ↑ De Vlaminck I., van Loenhout M. T., Zweifel L., den Blanken J., Hooning K., Hage S., Kerssemakers J., Dekker C. Mechanism of homology recognition in DNA recombination from dual-molecule experiments. (англ.) // Molecular cell. — 2012. — Vol. 46, no. 5. — P. 616—624. — doi:10.1016/j.molcel.2012.03.029. — PMID 22560720.
- ↑ Morimatsu K., Kowalczykowski S. C. RecFOR proteins load RecA protein onto gapped DNA to accelerate DNA strand exchange: a universal step of recombinational repair. (англ.) // Molecular cell. — 2003. — Vol. 11, no. 5. — P. 1337—1347. — PMID 12769856.
- ↑ 1 2 Handa N., Morimatsu K., Lovett S. T., Kowalczykowski S. C. Reconstitution of initial steps of dsDNA break repair by the RecF pathway of E. coli. (англ.) // Genes & development. — 2009. — Vol. 23, no. 10. — P. 1234—1245. — doi:10.1101/gad.1780709. — PMID 19451222.
- ↑ Hiom K. DNA repair: common approaches to fixing double-strand breaks. (англ.) // Current biology : CB. — 2009. — Vol. 19, no. 13. — P. 523—525. — doi:10.1016/j.cub.2009.06.009. — PMID 19602417.
- ↑ 1 2 West S. C. Molecular views of recombination proteins and their control. (англ.) // Nature reviews. Molecular cell biology. — 2003. — Vol. 4, no. 6. — P. 435—445. — doi:10.1038/nrm1127. — PMID 12778123.
- ↑ 1 2 3 4 James D. Watson, Tania A. Baker, Stephen P. Bell, Alexander Gann, Michael Levine, Richard Losick. Molecular Biology of the Gene (неопр.). — 5th. — Pearson/Benjamin Cummings, 2003. — С. 259—291. — ISBN 978-0-8053-4635-0.
- ↑ Gumbiner-Russo L. M., Rosenberg S. M. Physical analyses of E. coli heteroduplex recombination products in vivo: on the prevalence of 5' and 3' patches. (англ.) // Public Library of Science ONE. — 2007. — Vol. 2, no. 11. — P. e1242. — doi:10.1371/journal.pone.0001242. — PMID 18043749.
- ↑ Thomas C. M., Nielsen K. M. Mechanisms of, and barriers to, horizontal gene transfer between bacteria. (англ.) // Nature reviews. Microbiology. — 2005. — Vol. 3, no. 9. — P. 711—721. — doi:10.1038/nrmicro1234. — PMID 16138099.
- ↑ Vulić M., Dionisio F., Taddei F., Radman M. Molecular keys to speciation: DNA polymorphism and the control of genetic exchange in enterobacteria. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 1997. — Vol. 94, no. 18. — P. 9763—9767. — PMID 9275198.
- ↑ Majewski J., Cohan F. M. The effect of mismatch repair and heteroduplex formation on sexual isolation in Bacillus. (англ.) // Genetics. — 1998. — Vol. 148, no. 1. — P. 13—18. — PMID 9475717.
- ↑ Majewski J., Zawadzki P., Pickerill P., Cohan F. M., Dowson C. G. Barriers to genetic exchange between bacterial species: Streptococcus pneumoniae transformation. (англ.) // Journal of bacteriology. — 2000. — Vol. 182, no. 4. — P. 1016—1023. — PMID 10648528.
- ↑ Chen I., Dubnau D. DNA uptake during bacterial transformation. (англ.) // Nature reviews. Microbiology. — 2004. — Vol. 2, no. 3. — P. 241—249. — doi:10.1038/nrmicro844. — PMID 15083159.
- ↑ Claverys J. P., Martin B., Polard P. The genetic transformation machinery: composition, localization, and mechanism. (англ.) // FEMS microbiology reviews. — 2009. — Vol. 33, no. 3. — P. 643—656. — doi:10.1111/j.1574-6976.2009.00164.x. — PMID 19228200.
- ↑ Kidane D., Graumann P. L. Intracellular protein and DNA dynamics in competent Bacillus subtilis cells. (англ.) // Cell. — 2005. — Vol. 122, no. 1. — P. 73—84. — doi:10.1016/j.cell.2005.04.036. — PMID 16009134.
- ↑ Fleischmann Jr W. R. 43 // Medical Microbiology (неопр.). — 4th. — University of Texas Medical Branch at Galveston, 1996. — ISBN 0-9631172-1-1.
- ↑ Boni M. F., de Jong M. D., van Doorn H. R., Holmes E. C. Guidelines for identifying homologous recombination events in influenza A virus. (англ.) // Public Library of Science ONE. — 2010. — Vol. 5, no. 5. — P. e10434. — doi:10.1371/journal.pone.0010434. — PMID 20454662.
- ↑ 1 2 Nagy P. D., Bujarski J. J. Homologous RNA recombination in brome mosaic virus: AU-rich sequences decrease the accuracy of crossovers. (англ.) // Journal of virology. — 1996. — Vol. 70, no. 1. — P. 415—426. — PMID 8523555.
- ↑ Chetverin A. B. The puzzle of RNA recombination. (англ.) // FEBS letters. — 1999. — Vol. 460, no. 1. — P. 1—5. — PMID 10571050.
- ↑ 1 2 Roossinck M. J. Mechanisms of plant virus evolution. (англ.) // Annual review of phytopathology. — 1997. — Vol. 35. — P. 191—209. — doi:10.1146/annurev.phyto.35.1.191. — PMID 15012521.
- ↑ Arbuckle J. H., Medveczky P. G. The molecular biology of human herpesvirus-6 latency and telomere integration. (англ.) // Microbes and infection / Institut Pasteur. — 2011. — Vol. 13, no. 8-9. — P. 731—741. — doi:10.1016/j.micinf.2011.03.006. — PMID 21458587.
- ↑ Bernstein C. Deoxyribonucleic acid repair in bacteriophage. (англ.) // Microbiological reviews. — 1981. — Vol. 45, no. 1. — P. 72—98. — PMID 6261109.
- ↑ Bernstein C, Bernstein H. DNA repair in bacteriophage. In: Nickoloff JA, Hoekstra MF (Eds.) DNA Damage and Repair, Vol.3. Advances from Phage to Humans. Humana Press, Totowa, NJ. — 2001. — P. 1–19. — ISBN 978-0896038035.
- ↑ Story R. M., Bishop D. K., Kleckner N., Steitz T. A. Structural relationship of bacterial RecA proteins to recombination proteins from bacteriophage T4 and yeast. (англ.) // Science (New York, N.Y.). — 1993. — Vol. 259, no. 5103. — P. 1892—1896. — PMID 8456313.
- ↑ Michod R. E., Bernstein H., Nedelcu A. M. Adaptive value of sex in microbial pathogens. (англ.) // Infection, genetics and evolution : journal of molecular epidemiology and evolutionary genetics in infectious diseases. — 2008. — Vol. 8, no. 3. — P. 267—285. — doi:10.1016/j.meegid.2008.01.002. — PMID 18295550.
- ↑ Lamb N. E., Yu K., Shaffer J., Feingold E., Sherman S. L. Association between maternal age and meiotic recombination for trisomy 21. (англ.) // American journal of human genetics. — 2005. — Vol. 76, no. 1. — P. 91—99. — doi:10.1086/427266. — PMID 15551222.
- ↑ Cold Spring Harbor Laboratory. Human RecQ Helicases, Homologous Recombination And Genomic Instability. ScienceDaily (2007). Дата обращения: 3 июля 2010. Архивировано 10 сентября 2015 года.
- ↑ Modesti M., Kanaar R. Homologous recombination: from model organisms to human disease. (англ.) // Genome biology. — 2001. — Vol. 2, no. 5. — P. 1014. — PMID 11387040.
- ↑ Luo G., Santoro I. M., McDaniel L. D., Nishijima I., Mills M., Youssoufian H., Vogel H., Schultz R. A., Bradley A. Cancer predisposition caused by elevated mitotic recombination in Bloom mice. (англ.) // Nature genetics. — 2000. — Vol. 26, no. 4. — P. 424—429. — doi:10.1038/82548. — PMID 11101838.
- ↑ 1 2 3 Powell S. N., Kachnic L. A. Roles of BRCA1 and BRCA2 in homologous recombination, DNA replication fidelity and the cellular response to ionizing radiation. (англ.) // Oncogene. — 2003. — Vol. 22, no. 37. — P. 5784—5791. — doi:10.1038/sj.onc.1206678. — PMID 12947386.
- ↑ 1 2 3 4 5 6 Lin Z., Kong H., Nei M., Ma H. Origins and evolution of the recA/RAD51 gene family: evidence for ancient gene duplication and endosymbiotic gene transfer. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2006. — Vol. 103, no. 27. — P. 10328—10333. — doi:10.1073/pnas.0604232103. — PMID 16798872.
- ↑ 1 2 PMID 19282450
- ↑ Rolfsmeier M. L., Haseltine C. A. The single-stranded DNA binding protein of Sulfolobus solfataricus acts in the presynaptic step of homologous recombination. (англ.) // Journal of molecular biology. — 2010. — Vol. 397, no. 1. — P. 31—45. — doi:10.1016/j.jmb.2010.01.004. — PMID 20080104.
- ↑ Huang Q., Liu L., Liu J., Ni J., She Q., Shen Y. Efficient 5'-3' DNA end resection by HerA and NurA is essential for cell viability in the crenarchaeon Sulfolobus islandicus. (англ.) // BMC molecular biology. — 2015. — Vol. 16. — P. 2. — doi:10.1186/s12867-015-0030-z. — PMID 25880130.
- ↑ Jain S. K., Cox M. M., Inman R. B. On the role of ATP hydrolysis in RecA protein-mediated DNA strand exchange. III. Unidirectional branch migration and extensive hybrid DNA formation. (англ.) // The Journal of biological chemistry. — 1994. — Vol. 269, no. 32. — P. 20653—20661. — PMID 8051165.
- ↑ Ramesh M. A., Malik S. B., Logsdon J. M. Jr. A phylogenomic inventory of meiotic genes; evidence for sex in Giardia and an early eukaryotic origin of meiosis. (англ.) // Current biology : CB. — 2005. — Vol. 15, no. 2. — P. 185—191. — doi:10.1016/j.cub.2005.01.003. — PMID 15668177.
- ↑ 1 2 Malik S. B., Ramesh M. A., Hulstrand A. M., Logsdon J. M. Jr. Protist homologs of the meiotic Spo11 gene and topoisomerase VI reveal an evolutionary history of gene duplication and lineage-specific loss. (англ.) // Molecular biology and evolution. — 2007. — Vol. 24, no. 12. — P. 2827—2841. — doi:10.1093/molbev/msm217. — PMID 17921483.
- ↑ Lodish H., Berk A., Zipursky S. L., Matsudaira P., Baltimore D., Darnell J. Chapter 8.5: Gene Replacement and Transgenic Animals: DNA Is Transferred into Eukaryotic Cells in Various Ways // Molecular Cell Biology (неопр.). — 4th. — W. H. Freeman and Company, 2000. — ISBN 0-7167-3136-3.
- ↑ The Nobel Prize in Physiology or Medicine 2007. The Nobel Foundation. Дата обращения: 15 декабря 2008. Архивировано 8 декабря 2015 года.
- ↑ Masters J. R. Human cancer cell lines: fact and fantasy. (англ.) // Nature reviews. Molecular cell biology. — 2000. — Vol. 1, no. 3. — P. 233—236. — doi:10.1038/35043102. — PMID 11252900.
- ↑ Drummond D. A., Silberg J. J., Meyer M. M., Wilke C. O., Arnold F. H. On the conservative nature of intragenic recombination. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2005. — Vol. 102, no. 15. — P. 5380—5385. — doi:10.1073/pnas.0500729102. — PMID 15809422.
- ↑ Bloom J. D., Silberg J. J., Wilke C. O., Drummond D. A., Adami C., Arnold F. H. Thermodynamic prediction of protein neutrality. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2005. — Vol. 102, no. 3. — P. 606—611. — doi:10.1073/pnas.0406744102. — PMID 15644440.
- ↑ 1 2 Carbone M. N., Arnold F. H. Engineering by homologous recombination: exploring sequence and function within a conserved fold. (англ.) // Current opinion in structural biology. — 2007. — Vol. 17, no. 4. — P. 454—459. — doi:10.1016/j.sbi.2007.08.005. — PMID 17884462.
- ↑ Otey C. R., Landwehr M., Endelman J. B., Hiraga K., Bloom J. D., Arnold F. H. Structure-guided recombination creates an artificial family of cytochromes P450. (англ.) // Public Library of Science Biology. — 2006. — Vol. 4, no. 5. — P. e112. — doi:10.1371/journal.pbio.0040112. — PMID 16594730.
- ↑ Socolich M., Lockless S. W., Russ W. P., Lee H., Gardner K. H., Ranganathan R. Evolutionary information for specifying a protein fold. (англ.) // Nature. — 2005. — Vol. 437, no. 7058. — P. 512—518. — doi:10.1038/nature03991. — PMID 16177782.
- ↑ Thulasiram H. V., Erickson H. K., Poulter C. D. Chimeras of two isoprenoid synthases catalyze all four coupling reactions in isoprenoid biosynthesis. (англ.) // Science (New York, N.Y.). — 2007. — Vol. 316, no. 5821. — P. 73—76. — doi:10.1126/science.1137786. — PMID 17412950.
- ↑ Landwehr M., Carbone M., Otey C. R., Li Y., Arnold F. H. Diversification of catalytic function in a synthetic family of chimeric cytochrome p450s. (англ.) // Chemistry & biology. — 2007. — Vol. 14, no. 3. — P. 269—278. — doi:10.1016/j.chembiol.2007.01.009. — PMID 17379142.
- ↑ 1 2 3 Iglehart J. D., Silver D. P. Synthetic lethality--a new direction in cancer-drug development. (англ.) // The New England journal of medicine. — 2009. — Vol. 361, no. 2. — P. 189—191. — doi:10.1056/NEJMe0903044. — PMID 19553640.
- ↑ Fong P. C., Boss D. S., Yap T. A., Tutt A., Wu P., Mergui-Roelvink M., Mortimer P., Swaisland H., Lau A., O'Connor M. J., Ashworth A., Carmichael J., Kaye S. B., Schellens J. H., de Bono J. S. Inhibition of poly(ADP-ribose) polymerase in tumors from BRCA mutation carriers. (англ.) // The New England journal of medicine. — 2009. — Vol. 361, no. 2. — P. 123—134. — doi:10.1056/NEJMoa0900212. — PMID 19553641.
- ↑ Edwards S. L., Brough R., Lord C. J., Natrajan R., Vatcheva R., Levine D. A., Boyd J., Reis-Filho J. S., Ashworth A. Resistance to therapy caused by intragenic deletion in BRCA2. (англ.) // Nature. — 2008. — Vol. 451, no. 7182. — P. 1111—1115. — doi:10.1038/nature06548. — PMID 18264088.
Литература[править | править код]
- Б. Альбертс, А. Джонсон, Д. Льюис и др. Молекулярная биология клетки: в 3-х томах. — М. — Ижевск: НИЦ «Регулярная и хаотическая динамика», Институт компьютерных исследований, 2013. — Т. 1. — 808 с. — ISBN 978-5-4344-0112-8.
Ссылки[править | править код]
- Анимация – гомологичная рекомбинация: анимация, показывающая разные модели гомологичной рекомбинации.
- Гомологичная рекомбинация: Tempy & Trun: анимация бактериального механизма RecBCD в процессе ГР.
Эта статья входит в число избранных статей русскоязычного раздела Википедии. |


 French
French Deutsch
Deutsch