SARS-CoV – Wikipédia, a enciclopédia livre
| Coronavírus relacionado à síndrome respiratória aguda grave | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Classificação científica | |||||||||||||
| |||||||||||||
| Distribuição geográfica | |||||||||||||
 Mapa dos países infectados pelo SARS-CoV: Países com mortes confirmadas Países com casos confirmados Países sem casos confirmados | |||||||||||||
O coronavírus relacionado à síndrome respiratória aguda grave (SARS-CoV ou SARS-CoV-1), é o vírus que causa a síndrome respiratória aguda grave (SARS, conhecida como SRAG, em português).[1][2] Em 16 de abril de 2003, após o surto de SARS na Ásia e em casos secundários em outras partes do mundo, a Organização Mundial da Saúde (OMS) divulgou um comunicado de imprensa afirmando que o coronavírus identificado por vários laboratórios era a causa oficial da SARS. Amostras do vírus estão sendo mantidas em laboratórios na cidade de Nova York, São Francisco, Manila, Hong Kong e Toronto .
Em 12 de abril de 2003, os cientistas que trabalhavam no Centro de Ciências Genoma Michael Smith, em Vancouver, terminaram o mapeamento da sequência genética de um coronavírus que se acredita estar ligado à SARS. A equipe foi liderada pelo Dr. Marco Marra e trabalhou em colaboração com o Centro de Controle de Doenças da Columbia Britânica e o Laboratório Nacional de Microbiologia em Winnipeg, Manitoba, usando amostras de pacientes infectados em Toronto . O mapa, aclamado pela OMS como um importante passo à frente no combate à SARS, é compartilhado com cientistas em todo o mundo através do site do GSC (veja abaixo). O Dr. Donald Low, do Hospital Mount Sinai, em Toronto, descreveu a descoberta como tendo sido feita com "velocidade sem precedentes".[3] A sequência do coronavírus SARS foi confirmada por outros grupos independentes.
O coronavírus SARS é um dos vários vírus identificados pela OMS como causa provável de uma futura epidemia em um novo plano desenvolvido após a epidemia de Ebola para pesquisa e desenvolvimento urgentes antes e durante uma epidemia para novos testes de diagnóstico, vacinas e medicamentos.[4][5]
Em 2019 e 2020, outro vírus do mesmo grupo que o SARS-CoV, o SARS-CoV-2, se tornou responsável por causar a infecção covid-19 em humanos, causando a Pandemia de COVID-19.[6]
SARS[editar | editar código-fonte]
A SARS, ou síndrome respiratória aguda grave, é a doença causada pelo coronavírus SARS-CoV-1. Causa uma doença muitas vezes grave, marcada inicialmente por sintomas sistêmicos de dor muscular, dor de cabeça e febre, seguida em 2 a 14 dias pelo aparecimento de sintomas respiratórios,[7] principalmente tosse, dispneia e pneumonia . Outro achado comum em pacientes com SARS é uma diminuição no número de linfócitos que circulam no sangue.[8]
No surto de SARS de 2003, cerca de 9% dos pacientes com infecção confirmada por SARS morreram.[9] A taxa de mortalidade foi muito maior para aqueles com mais de 60 anos, com taxas de mortalidade próximas de 50% para esse subconjunto de pacientes.
História[editar | editar código-fonte]
O CDC e o Laboratório Nacional de Microbiologia do Canadá identificaram o genoma da SARS em abril de 2003.[10][11] Cientistas da Universidade Erasmus de Roterdã, na Holanda, demonstraram que o coronavírus da SARS cumpria os postulados de Koch, confirmando-o como agente causador. Nos experimentos, os macacos infectados com o vírus desenvolveram os mesmos sintomas que as vítimas humanas da SARS.[12]
No final de maio de 2003, estudos de amostras de animais selvagens vendidos como alimento no mercado local em Guangdong, China, descobriram que o coronavírus SARS poderia ser isolado de civetas de palmeiras mascaradas ( Paguma sp.), Mas os animais nem sempre exibiam sinais clínicos. A conclusão preliminar foi que o vírus da SARS atravessou a barreira xenográfica do civeta das palmeiras para os seres humanos e mais de 10.000 civetas de palma mascarados foram mortos na província de Guangdong. O vírus também foi encontrado posteriormente em cães-guaxinim ( Nyctereuteus sp.), Texugos de furão ( Melogale spp.) E gatos domésticos. Em 2005, dois estudos identificaram um número de coronavírus do tipo SARS em morcegos chineses.[13][14] A análise filogenética desses vírus indicou uma alta probabilidade de que o coronavírus SARS tenha se originado em morcegos e se espalhado para os seres humanos diretamente ou através de animais mantidos nos mercados chineses. Os morcegos não mostraram sinais visíveis de doença, mas são os prováveis reservatórios naturais de coronavírus do tipo SARS. No final de 2006, cientistas do Centro Chinês de Controle e Prevenção de Doenças da Universidade de Hong Kong e do Centro de Controle e Prevenção de Doenças de Guangzhou estabeleceram um vínculo genético entre o coronavírus SARS que aparece em civetas e humanos, confirmando as alegações de que a doença havia saltado entre espécies .[15]
Virologia[editar | editar código-fonte]
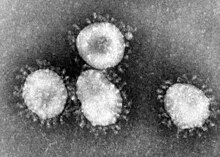
O coronavírus SARS é um vírus de RNA de cadeia simples e positivo, pertencente a uma família de coronavírus envolvidos. Seu genoma é de cerca de 29,7kb, um dos maiores entre os vírus de RNA. O vírus SARS possui 13 genes conhecidos e 14 proteínas conhecidas. Existem 265 nucleotídeos no 5'UTR e 342 nucleotídeos no 3'UTR. A SARS é semelhante a outros coronavírus, pois sua expressão do genoma começa com a tradução de duas ORFs grandes, 1a e 1b, ambas poliproteínas.
O receptor humano primário do vírus é a enzima conversora de angiotensina 2 (ACE2), identificada pela primeira vez em 2003.[16]
Morfologia[editar | editar código-fonte]
A morfologia do coronavírus SARS é característica da família dos coronavírus como um todo. Esses vírus possuem grandes partículas esféricas pleomórficas com projeções de superfície bulbosas que formam uma coroa em torno das partículas. O envelope do vírus contém lipídios e parece consistir em um par distinto de invólucros densos de elétrons.
Evolução[editar | editar código-fonte]
O SARS-CoV está mais relacionado aos coronavírus do grupo 2, mas não segrega em nenhum dos outros três grupos de coronavírus. Foi proposta uma teoria de que os coronavírus de morcegos foram co-evoluídos com seus hospedeiros por um longo tempo e depois saltaram espécies de morcegos para humanos.[17][18] O grupo externo mais próximo dos coronavírus são os torovírus, com os quais possui homologia na replicase ORF 1b e as duas proteínas viron de S e M. SARS foram determinadas como uma separação precoce dos coronavírus do grupo 2 com base em um conjunto de domínios compartilhados com o grupo 2.
Sintomas[editar | editar código-fonte]
Depois que uma pessoa contrai a SARS, o primeiro sintoma que ela apresenta é uma febre de pelo menos 38 ° C (100,4 ° F) ou superior. Os primeiros sintomas duram cerca de 2 a 7 dias e incluem sintomas inespecíficos do tipo gripe, incluindo calafrios / rigor, dores musculares, dores de cabeça, diarréia, dor de garganta, coriza, mal-estar e mialgia (dores musculares). Em seguida, desenvolvem tosse seca, falta de ar e infecção do trato respiratório superior.
O período de incubação da SARS-CoV é de 2 a 10 dias, às vezes durando até 13 dias, com média de 5 dias.[7] Assim, os sintomas geralmente se desenvolvem entre 2 a 10 dias após a infecção pelo vírus. Como parte da resposta imune, o anticorpo IgM para o SARS-CoV é produzido. Isso atinge o pico durante a fase convalescente aguda ou precoce (semana 3) e diminui na semana 12. O anticorpo IgG é produzido posteriormente e atinge o pico na semana 12.[19]
Projetando o vírus[editar | editar código-fonte]
A engenharia do vírus SARS foi realizada. Em um artigo publicado em 2006, um novo circuito de transcrição foi projetado para produzir vírus SARS recombinantes. A recombinação permitiu a expressão eficiente de transcritos virais e proteínas. A engenharia deste circuito de transcrição reduz os vírus da progênie recombinante de RNA. O circuito TRS (sequências reguladoras da transcrição) regula a expressão eficiente de mRNAs subgenômicos de SARS-CoV. O tipo selvagem TRS é ACGAAC.
Novas construções de vacina de subunidade para uma vacina contra a proteína S SARS, com base no domínio de ligação ao receptor (RBD, do inglês receptor-binding domain[20]), estão sendo desenvolvidas pelo New York Blood Center . O ressurgimento da SARS é possível e permanece a necessidade de vacinas comerciais e desenvolvimento terapêutico. No entanto, o custo e a duração do desenvolvimento do produto e a incerta demanda futura resultam em condições econômicas desfavoráveis para a realização dessa tarefa. No desenvolvimento de terapias e vacinas de próxima geração, é necessário mais trabalho para determinar as relações estrutura / função de enzimas críticas e proteínas estruturais.
Ver também[editar | editar código-fonte]
- Síndrome respiratória aguda grave
- Coronavírus da Síndrome Respiratória Aguda Grave 2 (SARS-CoV-2)
- COVID-19
Referências
- ↑ Thiel V (editor). (2007). Coronaviruses: Molecular and Cellular Biology. Caister Academic Press 1st ed. [S.l.: s.n.] ISBN 978-1-904455-16-5
- ↑ Lana, Raquel Martins; Coelho, Flávio Codeço; Gomes, Marcelo Ferreira da Costa; Cruz, Oswaldo Gonçalves; Bastos, Leonardo Soares; Villela, Daniel Antunes Maciel; Codeço, Cláudia Torres (2020). «Emergência do novo coronavírus (SARS-CoV-2) e o papel de uma vigilância nacional em saúde oportuna e efetiva». Cadernos de Saúde Pública. 36 (3): e00019620. ISSN 1678-4464. doi:10.1590/0102-311x00019620
- ↑ «B.C. lab cracks suspected SARS code». CBCNews, Canada
- ↑ «After Ebola, a Blueprint Emerges to Jump-Start R&D». Scientific American Blog Network
- ↑ «LIST OF PATHOGENS». World Health Organization
- ↑ Gobalenya AE, Baker SC, Baric RS, et al. (março de 2020). «The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2». Nature Microbiology: 1–9. PMID 32123347. doi:10.1038/s41564-020-0695-z
- ↑ a b «SARS: epidemiology». Respirology (Carlton, Vic.). 8: S9–14. PMID 15018127. doi:10.1046/j.1440-1843.2003.00518.x
- ↑ «Hematological findings in SARS patients and possible mechanisms (review)». International Journal of Molecular Medicine. 14: 311–5. PMID 15254784. doi:10.3892/ijmm.14.2.311
- ↑ «Severe acute respiratory syndrome (SARS): development of diagnostics and antivirals». Annals of the New York Academy of Sciences. 1067: 500–5. Bibcode:2006NYASA1067..500S. PMID 16804033. doi:10.1196/annals.1354.072
- ↑ «Remembering SARS: A Deadly Puzzle and the Efforts to Solve It». Centers for Disease Control and Prevention
- ↑ «Coronavirus never before seen in humans is the cause of SARS». United Nations World Health Organization
- ↑ «Aetiology: Koch's postulates fulfilled for SARS virus». Nature. 423. 2003. Bibcode:2003Natur.423..240F. PMID 12748632. doi:10.1038/423240a Verifique o valor de
|display-authors=Fouchier RA, Kuiken T, Schutten M, etal(ajuda) - ↑ «Bats are natural reservoirs of SARS-like coronaviruses». Science. 310: 676–9. 2005. Bibcode:2005Sci...310..676L. PMID 16195424. doi:10.1126/science.1118391 Verifique o valor de
|display-authors=Li W, Shi Z, Yu M, etal(ajuda) - ↑ «Severe acute respiratory syndrome coronavirus-like virus in Chinese horseshoe bats». Proc. Natl. Acad. Sci. U.S.A. 102: 14040–5. 2005. Bibcode:2005PNAS..10214040L. PMC 1236580
 . PMID 16169905. doi:10.1073/pnas.0506735102 Verifique o valor de
. PMID 16169905. doi:10.1073/pnas.0506735102 Verifique o valor de |display-authors=Lau SK, Woo PC, Li KS, etal(ajuda) - ↑ «Scientists prove SARS-civet cat link». China Daily
- ↑ Li. «Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus». Nature (em inglês). 426: 450–454. ISSN 0028-0836. PMID 14647384. doi:10.1038/nature02145
- ↑ «Evolutionary relationships between bat coronaviruses and their hosts». Emerg. Infect. Dis. 13: 1526–32. PMC 2851503
 . PMID 18258002. doi:10.3201/eid1310.070448
. PMID 18258002. doi:10.3201/eid1310.070448 - ↑ «Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor». Nature. 503: 535–8. Bibcode:2013Natur.503..535G. PMC 5389864
 . PMID 24172901. doi:10.1038/nature12711
. PMID 24172901. doi:10.1038/nature12711 - ↑ «Laboratory diagnosis of SARS». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 359: 1083–9. PMC 1693399
 . PMID 15306394. doi:10.1098/rstb.2004.1493
. PMID 15306394. doi:10.1098/rstb.2004.1493 - ↑ Tai, Wanbo; He, Lei; Zhang, Xiujuan; Pu, Jing; Voronin, Denis; Jiang, Shibo; Zhou, Yusen; Du, Lanying (19 de março de 2020). «Characterization of the receptor-binding domain (RBD) of 2019 novel coronavirus: implication for development of RBD protein as a viral attachment inhibitor and vaccine». Cellular & Molecular Immunology (em inglês): 1–8. ISSN 2042-0226. doi:10.1038/s41423-020-0400-4
Bibliografia[editar | editar código-fonte]
- «Coronavirus as a possible cause of severe acute respiratory syndrome». Lancet. 361: 1319–25. PMID 12711465. doi:10.1016/s0140-6736(03)13077-2 Verifique o valor de
|display-authors=Peiris JS, Lai ST, Poon LL, etal(ajuda) - «Characterization of a Novel Coronavirus Associated with Severe Acute Respiratory Syndrome». Science. 300: 1394–9. Bibcode:2003Sci...300.1394R. PMID 12730500. doi:10.1126/science.1085952 Verifique o valor de
|display-authors=Rota PA, Oberste MS, Monroe SS, etal(ajuda) - Marco A. Marra; et al. «The Genome Sequence of the SARS-Associated coronavirus». Science. 300: 1399–1404. Bibcode:2003Sci...300.1399M. PMID 12730501. doi:10.1126/science.1085953
- «Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage». J Mol Biol. 331: 991–1004. CiteSeerX 10.1.1.319.7007
 . PMID 12927536. doi:10.1016/S0022-2836(03)00865-9 Verifique o valor de
. PMID 12927536. doi:10.1016/S0022-2836(03)00865-9 Verifique o valor de |display-authors=Snijder EJ, etal(ajuda) - «Rewiring the severe acute respiratory syndrome coronavirus (SARS-CoV) transcription circuit: engineering a recombination-resistant genome». Proc Natl Acad Sci U S A. 103: 12546–51. Bibcode:2006PNAS..10312546Y. PMC 1531645
 . PMID 16891412. doi:10.1073/pnas.0605438103 Verifique o valor de
. PMID 16891412. doi:10.1073/pnas.0605438103 Verifique o valor de |display-authors=Yount B, etal(ajuda) - Thiel V, ed. (2007). Coronaviruses: Molecular and Cellular Biology. Caister Academic Press 1st ed. [S.l.: s.n.] ISBN 978-1-904455-16-5
- «Coronavirus Replication and Interaction with Host». Animal Viruses: Molecular Biology. Caister Academic Press. [S.l.: s.n.] 2008. ISBN 978-1-904455-22-6
Ligações externas[editar | editar código-fonte]
- Comunicado de imprensa da OMS identificando e nomeando o vírus SARS
- O mapa genético do vírus SARS
- Especial de ciências sobre o vírus SARS(conteúdo gratuito: não é necessário registro)
- McGill University SARS Resources no Wayback Machine (arquivado em março 1, 2005)
- Centros dos EUA para Controle e Prevenção de Doenças (CDC) SARS home
- Organização Mundial da Saúde em alerta


 French
French Deutsch
Deutsch
