Henipavirus — Wikipédia
Espèces de rang inférieur
- Sous-famille des Orthoparamyxovirinae (en)
- Genre Aquaparamyxovirus (en)
- Genre Ferlavirus (en)
- Genre Henipavirus
- virus de Cedar
- virus de Hendra
- virus de Kumasi
- virus de Langya
- virus de Mojiang
- virus Nipah
- Genre Morbillivirus
- Genre Narmovirus
- Genre Respirovirus (en)
- Genre Salemvirus
Henipavirus est un genre de virus de la famille des Paramyxoviridae[3], ordre des Mononegavirales, virus à ARN monocaténaire de polarité négative et à génome non segmenté. On y compte notamment le virus Hendra, initialement classé comme morbillivirus des équidés, et le virus Nipah, à l'origine d'épidémies d'infections à virus Nipah en Asie du Sud et du Sud-Est : le mot « henipavirus » est une contraction de ces deux noms de virus. Les virus du genre Henipavirus sont caractérisés par leur grande taille (18,2 kilobases[4]), leur occurrence naturelle chez les chauves-souris du genre Pteropus (roussettes), et leur émergence récente comme pathogène zoonotique potentiellement mortel chez les animaux domestiques et les humains. L'ARN de ces virus a également été identifié dans les déjections de roussettes paillées africaines au Ghana[5], indiquant la présence du virus en Afrique subsaharienne, dans la mesure où cette chauve-souris est très largement distribuée sur le continent.
Structure[modifier | modifier le code]
Les virions d’Henipavirus sont pléomorphes, c'est-à-dire qu'ils présentent des formes variables, avec une taille variant de 40 à 600 nm de diamètre[6]. Ils sont recouverts d'une enveloppe virale formée d'une bicouche lipidique recouvrant des protéines de matrice notées M. La surface du virus présente des glycoprotéines G assurant l'adhérence (attachment en anglais) du virus sur la cellule hôte, en particulier sur l'éphrine B2, une protéine hautement conservée présente chez de nombreux mammifères[7],[8], et des trimères de protéines F assurant la fusion de la membrane virale avec la membrane plasmique de la cellule hôte, un effet collatéral étant la formation dans grands syncytia résultant de la fusion de plusieurs cellules hôtes voisines sous l'effet de cette protéine virale[9] ; la structure tridimensionnelle de la protéine d'attachement G du virus Nipah a été établie par cristallographie aux rayons X[10]. L'intérieur du virus est constitué d'un ARN viral et d'une protéine N de nucléocapside complexée avec une protéine L fonctionnant comme enzyme de type ARN polymérase ARN-dépendante elle-même liée à une phosphoprotéine P indispensable à l'activité enzymatique de la protéine L.

Génome[modifier | modifier le code]
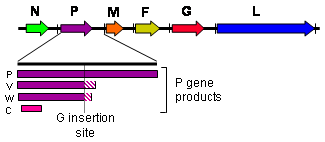
Le génome des Henipavirus est long de 18,2 kilobases et code six protéines structurelles[4]. Comme c'est la règle chez les paramyxovirus[13], le nombre de nucléotides dans ce génome est un multiple de six, ce qu'on appelle « la règle de six[14] ». Un écart par rapport à cette règle, par mutation ou formation d'un génome incomplet, rend inefficace la multiplication du virus[15], vraisemblablement du fait de contraintes structurelles dues à l'interaction entre l'ARN et la nucléoprotéine N du virus[16]. Ces virus expriment plusieurs protéines différentes à partir de leur gène P à la suite d'un processus d'édition des ARN messagers, dans ce cas par une modification post-transcriptionnelle consistant en l'ajout d'un ou deux résidus de guanosine au niveau d'un site d'édition du gène P avant sa transcription par les ribosomes de la cellule hôte[11] : le gène P produit ainsi une protéine P à partir de l'ARN messager non modifié, une protéine V dans le cas de l'insertion d'un résidu de guanosine, et une protéine W dans le cas de l'insertion de deux résidus de guanosine ; un quatrième produit de transcription, la protéine C, résulte d'un cadre de lecture ouvert alternatif. Si la phosphoprotéine P contribue à l'activité ARN polymérase ARN-dépendante de la protéine L, les protéines V, W et C jouent un rôle d'inhibiteur des défenses antivirales de la cellule hôte, sans que les mécanismes exacts de leur mode d'action soit bien compris[11],[12].
Virus de Hendra[modifier | modifier le code]
Apparition[modifier | modifier le code]
L'Hendravirus a été découvert en Australie en septembre 1994, quand il a causé la mort de trente chevaux et d'un entraîneur à Hendra, une banlieue de Brisbane dans le Queensland.
Le premier cas connu, une jument, est morte deux jours après être tombée malade. Elle était logée avec vingt-trois autres chevaux. Dix-neuf d'entre eux ont été contaminés et douze ont succombé. L'entraîneur et un garçon d'écurie qui avaient nourri la jument sont tombés malades moins d'une semaine après le décès de l'animal. Ils présentaient alors les symptômes de la grippe. Le garçon d'écurie a récupéré, mais l'entraîneur est mort de problèmes respiratoires et rénaux. Ils avaient été probablement contaminés par les éternuements de la jument.
Le deuxième cas était en fait antérieur. Le virus avait déjà frappé en , dans le nord de Mackay à 1 000 km de Brisbane, mais les deux n'ont été reliés que plus tard. L'infection a tué deux chevaux et leur propriétaire. Ce dernier a été admis à l'hôpital pour une méningite, trois semaines après avoir assisté à l'autopsie de ses chevaux. Il a récupéré, mais 14 mois plus tard, il développait des troubles neurologiques et mourait peu après. C'est la présence de l'Hendravirus dans le cerveau du patient qui a permis le diagnostic a posteriori.
À la suite d'analyses sériques de la faune dans les secteurs de manifestation du virus, il est apparu que les chauves-souris Pteropus étaient la source la plus probable de l'infection[17], avec une séroprévalence de 47 %. Aucune des 46 autres espèces n'était positive. La présence du virus dans l'appareil reproducteur et l'urine des chauve-souris permet de penser que les chevaux ont été contaminés par l'exposition à l'urine ou au liquide amniotique des chauve-souris.
Autres cas[modifier | modifier le code]
Trois cas supplémentaires sont signalés : deux à Cairns dans le Queensland, en et en , et un troisième à Townsville, toujours dans le Queensland, en . Chacun a eu comme conséquence la mort d'un cheval. Un vétérinaire ayant participé à l'autopsie du cheval de Townsville a développé une maladie lié à ce virus, mais a récupéré.

La distribution des chauve-souris correspond aux sites où des cas sont apparus. Par ailleurs, leur aspect saisonnier indiqueraient qu'ils sont liés au cycle de reproduction des Pteropus. Dans la mesure où il n'y a aucune preuve de transmission directe entre la chauve-souris et l'homme, on pense que celui-ci ne peut être infecté que par l'intermédiaire d'un hôte.
Pathologie[modifier | modifier le code]
Les chauves-souris ne sont pas affectées par le virus Hendra. Chez les humains, on observe des symptômes respiratoires (hémorragie et œdème pulmonaire) ou encéphaliques — dans ce cas, ils se traduisent par une méningite. Chez les chevaux, l'infection entraîne habituellement un œdème pulmonaire et une congestion nasale.
Virus Nipah[modifier | modifier le code]
Apparition[modifier | modifier le code]
Les premières épidémies liées au virus Nipah ont eu lieu à Singapour et en Malaisie en 1998. Cependant le virus Nipah a été identifié en 1999 où il a causé un cas de maladie neurologique et respiratoire aux fermes de porc en Malaisie péninsulaire, ayant pour résultat le décès de 115 personnes sur 265[18] et l'euthanasie d'un million de porcs[19]. À Singapour, onze cas dont un mortel se sont produits avec des ouvriers d'abattoir exposés aux porcs importés des fermes malaisiennes affectées.
Les réservoirs naturels du virus Nipah sont les chauves-souris frugivores du genre Pteropus. La déforestation en Asie du Sud-Est a entraîné la destruction de leur habitat. Elles ont donc migré afin de trouver de la nourriture, et se sont réfugiées vers les fermes où elles ont infecté la nourriture des porcs. En effet, le virus est contenu entre autres dans leur salive. Le porc contaminé excrète par de fortes toux le virus et contamine à son tour l'homme. Il est donc un hôte intermédiaire et aussi un puissant vecteur de l'infection.
Les symptômes de l'infection du cas malaisien étaient principalement encéphalitiques chez l'homme et respiratoires chez les porcs. Les manifestations postérieures ont causé des maladies respiratoires chez l'homme, augmentant la probabilité de la transmission humain-à-humain et indiquant l'existence des contraintes plus dangereuses du virus.
Autres cas[modifier | modifier le code]
Six cas supplémentaires de virus Nipah se sont produits depuis 1998, un en Inde et cinq au Bangladesh. Tous les emplacements de ces cas se trouvent en dessous de la zone des espèces de Pteropus (Pteropus giganteus). Comme avec le virus Hendra, la synchronisation des manifestations indique un effet saisonnier.
- - , Siliguri, Inde : 66 cas avec un taux de mortalité de 74 %[20]. 75 % de patients étaient soit le personnel d'hôpital, soit des personnes ayant rendu visite à d'autres patients dans l'hôpital, indiquant une transmission humain-à-humain.
- avril - , zone de Meherpur, Bangladesh : 13 cas avec neuf morts (mortalité de 69 %)[21].
- , zone de Naogaon, Bangladesh : 12 cas avec huit morts (mortalité de 67 %)[21].
- janvier - , provinces de Manikganj et de Rajbari, Bangladesh : 42 cas avec 14 morts (mortalité de 33 %).
- - , zone de Faridpur, Bangladesh : 36 cas avec 27 morts (mortalité de 75 %). Les indices épidémiologiques suggèrent fortement que ces cas aient impliqués la transmission de personne à personne du virus Nipah, qui n'avait pas été précédemment confirmée. 92 % de cas impliqués était étroitement en contact avec au moins une autre personne atteinte du virus Nipah. Deux cas ont comporté une exposition courte simple à un patient malade, y compris un conducteur de pousse-pousse qui a transporté un patient à l'hôpital. En outre, au moins six cas ont impliqué le SRAS qui n'a pas été rapporté précédemment pour la maladie de virus Nipah chez l'homme. Ce symptôme est susceptible d'avoir aidé la transmission humain-à-humaine par la grande dispersion de gouttelette lors des éternuements.
- , zone de Tangail, Bangladesh : 32 cas avec 12 morts (mortalité de 38 %). Le virus a pu avoir été contracté par la boisson de jus de datte souillé par les excréments ou la salive de chauves-souris.
Onze cas isolés d'encéphalite dus au virus Nipah ont été également référencés au Bangladesh depuis 2001.
Le virus Nipah a été isolé dans la chauve-souris de Lyle (Pteropus lylei) au Cambodge[22] et l'ARN viral a été trouvé dans l'urine et la salive de la chauve-souris de Lyle et des larves de Horsfield (Hipposideros larvartus) de Thaïlande[23]. Les cas cambodgiens montrent que le virus est identique à 98 % avec le virus causant les cas de 1998. On n'a observé aucune infection humaine ou d'autres espèces au Cambodge ou en Thaïlande.
En à Nouméa, Nouvelle-Calédonie, trois roussettes du parc forestier ont été diagnostiquées porteuses du virus Nipah[24]. Les animaux ont été euthanasiés[25].
Pathologie[modifier | modifier le code]
Chez l'humain, les infectés présentent de la fièvre, des maux de tête et de la somnolence. La toux, la douleur abdominale, la nausée, le vomissement, la faiblesse, les problèmes avec l'ingestion et la vision brouillée sont relativement communs. 1/4 des patients ont eu des malaises et environ 60 % sont devenus comateux et ont pu avoir besoin d'aide respiratoire. Les patients présentant des formes graves de la maladie ont développé de l'hypertension grave, une fréquence cardiaque et une température très élevées.
Le virus Nipah est également connu pour causer des encéphalites lors des rechutes. Parmi les cas malaisiens initiaux, un patient a présenté une encéphalite environ 53 mois après son infection initiale. Il n'y a aucun traitement définitif pour l'encéphalite de Nipah, indépendamment des mesures de support, telles que la ventilation mécanique et la prévention de l'infection secondaire. La ribavirine, un médicament antiviral, a été testée dans les infections malaisiennes et les résultats sont encourageants, cependant des études sont encore nécessaires.
Chez les autres animaux, particulièrement chez les porcs, le virus cause des syndromes respiratoires et neurologiques.
Causes d'apparition[modifier | modifier le code]
L'apparition des henipavirus s'est faite en parallèle de l'apparition d'autres virus zoonotiques dans les décennies récentes. Les virus responsables du SRAS et de la maladie à virus Ebola (entre autres) ont aussi été probablement 'hébergés' par des chauves-souris et sont capables d'infecter une variété d'autres espèces. L'apparition de chacun de ces virus a été liée à une augmentation des contacts entre les chauves-souris et les humains, impliquant parfois un hôte intermédiaire. Le contact accru est conduit par l’empiétement humain dans le territoire des chauves-souris (dans le cas de Nipah, les élevages porcins sont dans ledit territoire) et par le mouvement des chauves-souris vers les populations humaines dû aux changements dans l'habitude alimentaire et à la perte d'habitat.
Il est évident que la perte d'habitat des chauves-souris en Asie du sud et en Australie (en particulier le long de la côte est) aussi bien que l'empiétement des logements humains et de l'agriculture dans les habitats restants, vont créer un plus grand contact entre les hommes et les chauves-souris.
Notes et références[modifier | modifier le code]
- (en) « Hendra Virus », CSIRO (consulté le ).
- ICTV. International Committee on Taxonomy of Viruses. Taxonomy history. Published on the Internet https://talk.ictvonline.org/., consulté le 5 février 2021
- (en) Gaya K. Amarasinghe, María A. Ayllón, Yīmíng Bào, Christopher F. Basler, Sina Bavari, Kim R. Blasdell, Thomas Briese, Paul A. Brown, Alexander Bukreyev, Anne Balkema-Buschmann, Ursula J. Buchholz, Camila Chabi-Jesus, Kartik Chandran, Chiara Chiapponi, Ian Crozier, Rik L. de Swart, Ralf G. Dietzgen, Olga Dolnik, Jan F. Drexler, Ralf Dürrwald, William G. Dundon, W. Paul Duprex, John M. Dye, Andrew J. Easton, Anthony R. Fooks, Pierre B. H. Formenty, Ron A. M. Fouchier, Juliana Freitas-Astúa, Anthony Griffiths, Roger Hewson, Masayuki Horie, Timothy H. Hyndman, Dàohóng Jiāng, Elliott W. Kitajima, Gary P. Kobinger, Hideki Kondō, Gael Kurath, Ivan V. Kuzmin, Robert A. Lamb, Antonio Lavazza, Benhur Lee, Davide Lelli, Eric M. Leroy, Jiànróng Lǐ, Piet Maes, Shin-Yi L. Marzano, Ana Moreno, Elke Mühlberger, Sergey V. Netesov, Norbert Nowotny, Are Nylund, Arnfinn L. Økland, Gustavo Palacios, Bernadett Pályi, Janusz T. Pawęska, Susan L. Payne, Alice Prosperi, Pedro Luis Ramos-González, Bertus K. Rima, Paul Rota, Dennis Rubbenstroth, Mǎng Shī, Peter Simmonds, Sophie J. Smither, Enrica Sozzi, Kirsten Spann, Mark D. Stenglein, David M. Stone, Ayato Takada, Robert B. Tesh, Keizō Tomonaga, Noël Tordo, Jonathan S. Towner, Bernadette van den Hoogen, Nikos Vasilakis, Victoria Wahl, Peter J. Walker, Lin-Fa Wang, Anna E. Whitfield, John V. Williams, F. Murilo Zerbini, Tāo Zhāng, Yong-Zhen Zhang et Jens H. Kuhn, « Taxonomy of the order Mononegavirales: update 2019 », Archives of Virology, vol. 164, no 7, , p. 1967-1980 (PMID 31089958, DOI 10.1007/s00705-019-04247-4, lire en ligne)
- (en) Lin-Fa Wang, Brian H. Harcourt, Meng Yu, Azaibi Tamin, Paul A. Rota, William J. Bellini et Bryan T. Eaton, « Molecular biology of Hendra and Nipah viruses », Microbes and Infection, vol. 3, no 4, , p. 279-287 (PMID 11334745, DOI 10.1016/S1286-4579(01)01381-8, lire en ligne)
- (en) Jan Felix Drexler, Victor Max Corman, Florian Gloza-Rausch, Antje Seebens, Augustina Annan, Anne Ipsen, Thomas Kruppa, Marcel A. Müller, Elisabeth K. V. Kalko, Yaw Adu-Sarkodie, Samuel Oppong et Christian Drosten, « Henipavirus RNA in African Bats », PLoS One, vol. 4, no 7, , article no e6367 (PMID 19636378, PMCID 2712088, DOI 10.1371/journal.pone.0006367, Bibcode 2009PLoSO...4.6367D, lire en ligne)
- (en) Alex D. Hyatt, Sherif R. Zaki, Cynthia S. Goldsmith, Terry G. Wise et Sandra G. Hengstberger, « Ultrastructure of Hendra virus and Nipah virus within cultured cells and host animals », Microbes and Infection, vol. 3, no 4, , p. 297-306 (PMID 11334747, DOI 10.1016/S1286-4579(01)01383-1, lire en ligne)
- (en) Matthew I. Bonaparte, Antony S. Dimitrov, Katharine N. Bossart, Gary Crameri, Bruce A. Mungall, Kimberly A. Bishop, Vidita Choudhry, Dimiter S. Dimitrov, Lin-Fa Wang, Bryan T. Eaton et Christopher C. Broder, « Ephrin-B2 ligand is a functional receptor for Hendra virus and Nipah virus », Proceedings of the National Academy of Sciences of the United States of America, vol. 102, no 30, , p. 10652-10657 (PMID 15998730, PMCID 1169237, DOI 10.1073/pnas.0504887102, JSTOR 3376138, Bibcode 2005PNAS..10210652B, lire en ligne)
- (en) Thomas A. Bowden, Max Crispin, E. Yvonne Jones et David I. Stuart, « Shared paramyxoviral glycoprotein architecture is adapted for diverse attachment strategies », Biochemical Society Transactions, vol. 38, no 5, , p. 1349-1355 (PMID 20863312, PMCID 3433257, DOI 10.1042/BST0381349, lire en ligne)
- (en) K. B. Chua, W. J. Bellini, P. A. Rota, B. H. Harcourt, A. Tamin, S. K. Lam, T. G. Ksiazek, P. E. Rollin, S. R. Zaki, W.-J. Shieh, C. S. Goldsmith, D. J. Gubler, J. T. Roehrig, B. Eaton, A. R. Gould, J. Olson, H. Field, P. Daniels, A. E. Ling, C. J. Peters, L. J. Anderson2 et B. W. J. Mahy, « Nipah Virus: A Recently Emergent Deadly Paramyxovirus », Science, vol. 288, no 5470, , p. 1432-1435 (PMID 10827955, DOI 10.1126/science.288.5470.1432, JSTOR 3075321, Bibcode 2000Sci...288.1432C, lire en ligne)
- (en) Thomas A. Bowden, Max Crispin, David J. Harvey, A. Radu Aricescu, Jonathan M. Grimes, E. Yvonne Jones et David I. Stuart, « Crystal Structure and Carbohydrate Analysis of Nipah Virus Attachment Glycoprotein: a Template for Antiviral and Vaccine Design », Journal of Virology, vol. 82, no 23, , p. 11628-11636 (PMID 18815311, PMCID 2583688, DOI 10.1128/JVI.01344-08, lire en ligne)
- (en) Sachin Kulkarni, Valentina Volchkova, Christopher F. Basler, Peter Palese, Viktor E. Volchkov et Megan L. Shaw, « Nipah Virus Edits Its P Gene at High Frequency To Express the V and W Proteins », Journal of Virology, vol. 83, no 8, , p. 3982-3987 (PMID 19211754, PMCID 2663244, DOI 10.1128/JVI.02599-08, lire en ligne)
- (en) Megan L. Shaw, Adolfo García-Sastre, Peter Palese et Christopher F. Basler, « Nipah Virus V and W Proteins Have a Common STAT1-Binding Domain yet Inhibit STAT1 Activation from the Cytoplasmic and Nuclear Compartments, Respectively », Journal of Virology, vol. 78, no 11, , p. 5633-5641 (PMID 15140960, PMCID 15140960, DOI 10.1128/JVI.78.11.5633-5641.2004, lire en ligne)
- (en) Daniel Kolakofsky, Thierry Pelet, Dominique Garcin, Stéphane Hausmann, Joseph Curran et Laurent Roux, « Paramyxovirus RNA Synthesis and the Requirement for Hexamer Genome Length: the Rule of Six Revisited », Journal of Virology, vol. 72, no 2, , p. 891-899 (PMID 9444980, PMCID 124558, lire en ligne)
- (en) Kim Halpin et Bruce A. Mungall, « Recent progress in henipavirus research », Comparative Immunology, Microbiology and Infectious Diseases, vol. 30, nos 5-6, , p. 287-307 (PMID 17629946, DOI 10.1016/j.cimid.2007.05.008, lire en ligne)
- (en) Kim Halpin, Bettina Bankamp, Brian H. Harcourt, William J. Bellini et Paul A. Rota, « Nipah virus conforms to the rule of six in a minigenome replication assay », The Journal of General Virology, vol. 85, no Pt 3, , p. 701-707 (PMID 14993656, DOI 10.1099/vir.0.19685-0, lire en ligne)
- (en) Filip Yabukarski, Philip Lawrence, Nicolas Tarbouriech, Jean-Marie Bourhis, Elise Delaforge, Malene Ringkjøbing Jensen, Rob W H Ruigrok, Martin Blackledge, Viktor Volchkov et Marc Jamin, « Structure of Nipah virus unassembled nucleoprotein in complex with its viral chaperone », Nature Structural & Molecular Biology, vol. 21, no 9, , p. 754-759 (PMID 25108352, DOI 10.1038/nsmb.2868, lire en ligne)
- Halpin et al. 2000
- OMS, WHO Director-General addresses the Asian Investment Conference Dr Margaret Chan Directrice générale OMS
- Field et al. 2001
- Chadha et al. 2006
- Hsu et al. 2004
- Reynes et al. 2005
- Wacharapluesadee et al. 2005
- « Le virus Nipah sur des roussettes du parc forestier », sur Les Nouvelles Calédoniennes, (consulté le )
- « Découverte du virus NIPAH en Nouvelle-Calédonie », sur La Province Sud, (consulté le )
Voir aussi[modifier | modifier le code]
Références biologiques[modifier | modifier le code]
- (en) Référence ICTV : Henipavirus (consulté le )
- (en) Référence NCBI : Henipavirus (taxons inclus) (consulté le )
Bibliographie[modifier | modifier le code]
![]() : document utilisé comme source pour la rédaction de cet article.
: document utilisé comme source pour la rédaction de cet article.
- (en) Field H, Young P, Yob JM, Mills J, Hall L. & Mackenzie J., « The natural history of Hendra and Nipah viruses », Microbes and Infection, vol. 3, no 4, , p. 307-14. (PMID 11334748)

- (en) Halpin K, Young PL, Filed HE, Mackenzie JS., « Isolation of Hendra virus from pteropid bats: a natural reservoir of Hendra virus », Journal of General Virology, vol. 81, no 8, , p. 1927-32. (PMID 10900029, lire en ligne)

- (en) Hsu VP, Hossain MJ, Parashar UD, Ali MM, Ksiazek TG, Kuzmin I, Niezgoda, M, Rupprecht C, Bresee J. & Brieman RF., « Nipah virus encephalitis reemergence, Bangladesh », Emerging Infectious Disease, vol. 10, no 12, , p. 2082-87. (PMCID PMC3323384, DOI 10.3201/eid1012.040701, lire en ligne)

- Chadha, MS, James, AC, Lowe, L. et al., « Nipah virus-associated encephalitis outbreak, Siliguri, India », Emerging Infectious Diseases, vol. 12, no 2, , p. 235-40. (PMID 16494748, PMCID PMC3373078, DOI 10.3201/eid1202.051247, lire en ligne)

- (en) Reynes JM, Counor D, Ong S, Faure C, Seng V, Molia S. et al., « Nipah virus in Lyle’s flying foxes, Cambodia », Emerging Infectious Diseases, vol. 11, no 7, (PMID 16022778, PMCID PMC3371782, DOI 10.3201/eid1107.041350, lire en ligne)

- (en) Wacharapluesadee S, Lumlertdacha B, Boongird K, Wanghongsa S, Chanhome L, Rollin P. et al., « Bat Nipah virus, Thailand », Emerging Infectious Diseases, vol. 11, no 12, , p. 1949-51 (PMID 16485487, PMCID PMC3367639, lire en ligne)

- (en) Wang L, Harcourt BH, Yu M, Tamin A, Rota PA, Bellini WJ. et Eaton BT., « Molecular biology of Hendra and Nipah viruses », Microbes and Infection, vol. 3, no 4, , p. 279-87. (PMID 11334745)

- (en) L.-F. Wang, « Henipaviruses », dans B.T. Eaton et L.-F. Wang, Encyclopedia of Virology, (DOI 10.1016/B978-0-12-801238-3.02579-4), p. 321-327
Liens externes[modifier | modifier le code]
- (en) Hendra virus CSIRO FAQ
- (en) Nipah virus CSIRO FAQ


 French
French Deutsch
Deutsch