Coronavirus murin — Wikipédia
| Groupe | (+)ssRNA |
|---|---|
| Ordre | Nidovirales |
| Famille | Coronaviridae |
| Sous-famille | Orthocoronavirinae |
| Genre | Betacoronavirus |
| Sous-genre | Embecovirus |
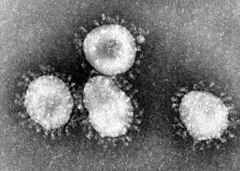
Le coronavirus murin (MCoV), connu avant 2009 sous le nom de virus de l’hépatite murine (MHV pour murine hepatitis virus en anglais), aussi appelé virus de la gastroentérite murine (murine gastroenteritis virus en anglais), est un coronavirus à ARN qui est responsable d’une hépatite chez la souris.
C’est le pathogène le plus important des souris de laboratoire, responsable d’épidémies dans cette population. Bien que l’infection soit la plupart du temps asymptomatique chez les souris adultes, elle est responsable de modifications du système immunitaire qui peuvent perturber les résultats scientifiques, raison pour laquelle les souris utilisées en expérimentation animale doivent en être indemnes.
Ce virus fait partie du groupe 2 des coronavirus. Vingt-cinq souches différentes ont été identifiées, les plus importantes étant MHV 1, MHV 2, MHV 3 et MHV 4. MHV 3 est responsable de l'hépatite la plus importante et est donc utilisé pour étudier les mécanismes immunitaires d'une hépatite virale chez la souris. MHV 4, également appelé MHV-JHM, a quant à lui un tropisme neurologique. Cette souche, responsable d’une encéphalite démyélinisante progressive, est utilisée comme modèle murin de la sclérose en plaques.
Position phylogénétique[modifier | modifier le code]
Le virus est un betacoronavirus, renommé « Murine coronavirus » et élevé au rang d'espèce en 2009. Depuis 2018, il est classé dans le sous-genre Embecovirus.
| β-CoV[2] |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Références[modifier | modifier le code]
- (en) « Virus Taxonomy: 2019 Release », ICTV, (consulté le ).
- DOI 10.1186/s44149-021-00005-9
- DOI 10.1128/JVI.01600-13
- DOI 10.1073/pnas.0506735102
- Shin Murakami, Tomoya Kitamura, Jin Suzuki, Ryouta Sato, Toshiki Aoi, Marina Fujii, Hiromichi Matsugo, Haruhiko Kamiki, Hiroho Ishida, Akiko Takenaka-Uema, Masayuki Shimojima et Taisuke Horimoto, « Detection and Characterization of Bat Sarbecovirus Phylogenetically Related to SARS-CoV-2, Japan », Emerging Infectious Diseases, vol. 26, no 12, , p. 3025–3029 (DOI 10.3201/eid2612.203386)
- Tommy Tsan-Yuk Lam, Na Jia, Ya-Wei Zhang, Marcus Ho-Hin Shum, Jia-Fu Jiang, Hua-Chen Zhu, Yi-Gang Tong, Yong-Xia Shi, Xue-Bing Ni, Yun-Shi Liao, Wen-Juan Li, Bao-Gui Jiang, Wei Wei, Ting-Ting Yuan, Kui Zheng, Xiao-Ming Cui, Jie Li, Guang-Qian Pei, Xin Qiang, William Yiu-Man Cheung, Lian-Feng Li, Fang-Fang Sun, Si Qin, Ji-Cheng Huang, Gabriel M. Leung, Edward C. Holmes, Yan-Ling Hu, Yi Guan et Wu-Chun Cao, « Identifying SARS-CoV-2-related coronaviruses in Malayan pangolins », Nature, vol. 583, no 7815, , p. 282–285 (DOI 10.1038/s41586-020-2169-0)
- Ping Liu, Jing-Zhe Jiang, Xiu-Feng Wan, Yan Hua, Linmiao Li, Jiabin Zhou, Xiaohu Wang, Fanghui Hou, Jing Chen, Jiejian Zou et Jinping Chen, « Are pangolins the intermediate host of the 2019 novel coronavirus (SARS-CoV-2)? », PLOS Pathogens, vol. 16, no 5, , e1008421 (DOI 10.1371/journal.ppat.1008421)
- (en) Vibol Hul, Deborah Delaune, Erik A. Karlsson, Alexandre Hassanin, Putita Ou Tey, Artem Baidaliuk, Fabiana Gámbaro, Vuong Tan Tu, Lucy Keatts, Jonna Mazet, Christine Johnson, Philippe Buchy, Philippe Dussart, Tracey Goldstein, Etienne Simon-Lorière et Veasna Duong, « A novel SARS-CoV-2 related coronavirus in bats from Cambodia », sur bioRxiv, (DOI 10.1101/2021.01.26.428212), p. 2021.01.26.428212
- S Wacharapluesadee, CW Tan, P Maneeorn, P Duengkae, F Zhu, Y Joyjinda, T Kaewpom, WN Chia, W Ampoot, BL Lim, K Worachotsueptrakun, VC Chen, N Sirichan, C Ruchisrisarod, A Rodpan, K Noradechanon, T Phaichana, N Jantarat, B Thongnumchaima, C Tu, G Crameri, MM Stokes, T Hemachudha et LF Wang, « Evidence for SARS-CoV-2 related coronaviruses circulating in bats and pangolins in Southeast Asia. », Nature Communications, vol. 12, no 1, , p. 972 (PMID 33563978, PMCID 7873279, DOI 10.1038/s41467-021-21240-1)
- H Zhou, X Chen, T Hu, J Li, H Song, Y Liu, P Wang, D Liu, J Yang, EC Holmes, AC Hughes, Y Bi et W Shi, « A Novel Bat Coronavirus Closely Related to SARS-CoV-2 Contains Natural Insertions at the S1/S2 Cleavage Site of the Spike Protein. », Current biology : CB, vol. 30, no 11, , p. 2196-2203.e3 (PMID 32416074, DOI 10.1016/j.cub.2020.05.023)
- « Addendum: A pneumonia outbreak associated with a new coronavirus of probable bat origin », Nature, vol. 588, no 7836, , E6 (PMID 33199918, DOI 10.1038/s41586-020-2951-z, lire en ligne)
- « Mouse Hepatitis Virus (MHV) », Division of Animal Resources, University of Illinois, Urbana (fiche de synthèse en anglais)


 French
French Deutsch
Deutsch