Receptor de insulina , la enciclopedia libre
| Receptor de insulina | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | Lista de códigos PDB | |||
| Identificadores | ||||
| Símbolo | INSR (HGNC: 6091) | |||
| Identificadores externos | Bases de datos de enzimas | |||
| Número EC | 2.7.10.1 | |||
| Locus | Cr. 19 p13.3-p13.2 | |||
| Patrón de expresión de ARNm | ||||
 | ||||
| Más información | ||||
| Estructura/Función proteica | ||||
| Tamaño | 1.382 (aminoácidos) | |||
| Peso molecular | 156.333 (Da) | |||
| Ortólogos | ||||
| Especies |
| |||
| Entrez |
| |||
| Ensembl |
| |||
| UniProt |
| |||
| RefSeq (ARNm) |
| |||
| RefSeq (proteína) NCBI |
| |||
| Ubicación (UCSC) |
| |||
| PubMed (Búsqueda) |
| |||
En biología molecular, el receptor de insulina o receptor insulínico[1] es un receptor transmembrana que es activado por la hormona insulina y pertenece a la familia de receptores de factor de crecimiento. El receptor es una glucoproteína, y es una enzima que pertenece al grupo de los receptores tirosina quinasas (EC 2.7.10.1). Este receptor en humanos se encuentra codificado por el gen INSR.[2]
La disfunción del receptor está relacionada con patologías graves como la diabetes mellitus, el cáncer y la enfermedad de Alzheimer.
Estructura[editar]

Derecha: modelo estructural.
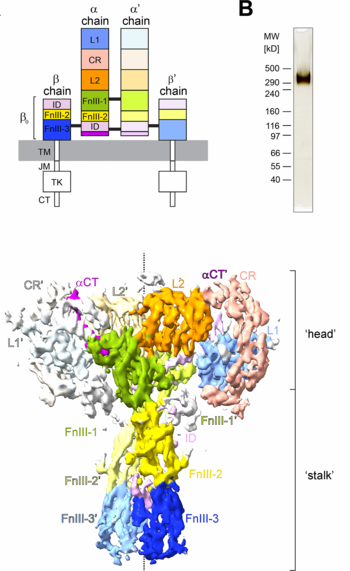
El receptor de insulina (INS-R) es una glucoproteína de la membrana plasmática, compuesta por cuatro subunidades (heterotetramero).
El receptor INS-R se puede dividir en dos dominios funcionales, las subunidades alfa (extracelulares) que se unen a la insulina y las subunidades beta (intracelulares) que poseen actividad de tirosina quinasa.[3]
Este receptor glucoproteico INS-R muestra actividad enzimática, al catalizar la transferencia de un grupo fosfato desde el ATP, a un aminoácido tirosina de la cadena proteica, por lo que pertenece al grupo de las receptores tirosina quinasas, (Anexo: EC 2.7.10.1).
Subunidades[editar]
TM= transmembrana; JM= yuxtamembrana; TK= dominio tirosina quinasa.
El receptor INS-R está conformado por dos subunidades alfa y dos subunidades beta (2 homo-dímeros α-β covalentes o como un hetero-tetrámero) con atividad quinasa.[4][5]
Ambos tipos de subunidades son sintetizadas de un precursor (pro-receptor) único, codificado por un gen localizado en el cromosoma 19 (humano). El transcripto de 1382 aminoácidos, sufre glucosilación, acilación y posterior proteólisis en el aparato de Golgi para generar el receptor definitivo con sus cuatro subunidades.[6] La proteína sintetizada es fraccionada (escisión proteolítica) en dos partes formando una subunidad alfa y una subunidad beta.
Dos de los sitios de unión a la insulina están en la región de la "cabeza" del receptor, los dos sitios adicionales para moléculas de insulina, están ubicados en las regiones del "tallo" de los dominios ECD (ectodominio o extracelular).
Subunidades alfa[editar]
Glicoproteína con masa de ~125 000 daltons (Da) para la subunidad alfa.[7][4]
Las dos subunidades alfa (α) se encuentran en el lado extracelular de la membrana. Las subunidades α se unen entre sí por puentes disulfuro, pero también están unidas a las subunidades beta por enlaces disulfuro, de modo que la molécula forma un solo complejo heterotetramérico.[8]
Las investigaciones bioquímicas predijeron uno o dos sitios de unión a la insulina: 1 y 2. La unión a la insulina estabiliza el ectodominio del receptor, en una conformación en forma de T, en la que los dominios próximos a la membrana convergen y se ponen en contacto entre sí. La unión de insulina al ectodominio eleva concomitantemente la actividad de tirosina quinasa intrínseca del receptor INS-R, antes de la transducción de la señal celular.
Estudios subsecuentes calcularon conformaciones del INS-R para tres moléculas y cuatro moléculas de insulina en otros tantos sitios de unión.[9][10][11]
Subunidades beta[editar]
Glicoproteína con masa de ~90 000 daltons (Da).[7]
Las dos subunidades beta (β) se insertan en la membrana celular y están unidas a las subunidades alfa mediante enlaces de disulfuro.
Las subunidades β tienen: una pequeña porción extracelular (ECD), una porción transmembranal (TM) y una porción intracelular (o intracitoplásmica) que posee la actividad tirosina quinasa entre los aminoácidos 1023-1298 de la cadena.[6]
Genética[editar]
El receptor de insulina es una proteína codificada por el gen INSR localizado en el cromosoma 19 (humano), brazo corto, banda p13.3-13.2.[12] Codifica una cadena de 1382 aminoácidos, que es clivada en 2 cadenas: la Insulin receptor subunidad alfa α y la Insulin receptor subunidad beta β.
Localización[editar]
Los tejidos con mayor abundancia de receptores de insulina son el parénquima hepático y el tejido adiposo, donde pueden llegar a existir 200 000 a 300 000 ( 3 × 105) copias del receptor por célula.[6]
Actividad[editar]

En el momento en que la molécula de insulina se une al receptor de insulina (INS-R), se produce un cambio en la conformación de la estructura de las subunidades del receptor. La unión de la insulina (ligando) estabiliza el ectodominio del INS-R, en una conformación en forma de T, en la que los dominios próximos a la membrana convergen y se ponen en contacto entre sí.[10]

El receptor de insulina transfiere grupos fosfato desde el ATP a ciertas proteínas específicas dentro de la célula. Esto conlleva un incremento del transporte de moléculas de glucosa desde la circulación sanguínea al interior de los miocitos y los adipocitos, y por lo tanto un incremento en la concentración de glucosa en el interior de las células del músculo y del tejido adiposo. La activación de este proceso bioquímico se logra en el momento en que una molécula de insulina se une al receptor, lo que induce la iniciación de las actividades enzimáticas en el interior de la célula.[4]
Véase también[editar]
Enlaces externos[editar]
Referencias[editar]
- ↑ OMS,OPS,BIREME (ed.). «Receptor de insulina». Descriptores en Ciencias de la Salud. Biblioteca Virtual en Salud.
- ↑ INSR «P06213·INSR_HUMAN». UniProt.
- ↑ Gammeltoft S.; Van Obberghen E. (1986). «Protein kinase activity of the insulin receptor». Biochem J. (REVISIÓN) 235 (1): 1-11. PMC 1146640. PMID 3017297. doi:10.1042/bj2350001. Consultado el 13 de mayo de 2023.

- ↑ a b c Carvalheira J.B.C.; Zecchin H.G.; Saad M.J.A. (2002). «Vias de Sinalização da Insulina.». Arq Bras Endocrinol Metab. (SciELO) 46 (4): 419-425. ISSN 0004-2730. doi:10.1590/S0004-27302002000400013. Consultado el 2 de enero de 2008.

- ↑ Gutmann T.; Kim K.H.; Grzybek M.; Walz T.; Coskun Ü. (2018). «Visualization of ligand-induced transmembrane signaling in the full-length human insulin receptor». J Cell Biol. Collection: The Year in Cell Biology:2018 217 (5): 1643-1649. Consultado el 6 de mayo de 2023.

- ↑ a b c Mendivil Anaya C.O.; Sierra Ariza I.D. (2005). «Acción insulínica y resistencia a la Insulina: aspectos moleculares». rev.fac.med. (Bogotá: SciELO) 53 (4). Consultado el 27 de enero de 2023.

- ↑ a b Czech M.P. (1985). «The nature and regulation of the insulin receptor: structure and function» (PDF). Annu Rev Physiol (REVISIÓN) (en inglés) 47: 357-381. PMID 2986534. doi:10.1146/annurev.ph.47.030185.002041. Consultado el 12 de mayo de 2023.

- ↑ Farmacología molecular: receptores «Receptor de insulina.». Instituto Químico Biológico. (IQB). Consultado el 2 de enero de 2007.
- ↑ Emiko Uchikawa; Eun Hee Choi; Guijun Shang; Hongtao Yu; Xiao Chen Bai (2019). «Activation mechanism of the insulin receptor revealed by cryo-EM structure of the fully liganded receptor–ligand complex». Elife. Structural Biology and Molecular Biophysics (en inglés) 8: e48630. doi:10.7554/eLife.48630. Consultado el 13 de mayo de 2023.

- ↑ a b Gutmann T.; Schäfer I.G.; Poojari C.; Brankatschk B.; Vattulainen I.; Strauss M.; Coskun U. (2020). «Cryo-EM structure of the complete and ligand-saturated insulin receptor ectodomain.». J Cell Biol 219 (1): e201907210. PMC 7039211. PMID 31727777. doi:10.1083/jcb.201907210.

- ↑ Nielsen J.; Brandt J.; Boesen T.; Hummelshøj T.; Slaaby R.; Schluckebier G.; Nissen P. (2022). «Structural Investigations of Full-Length Insulin Receptor Dynamics and Signalling». J Mol Biol (en inglés) 434 (5): 167458. PMID 35074483. doi:10.1016/j.jmb.2022.167458. Consultado el 13 de mayo de 2023.

- ↑ «P06213· INSR_HUMAN». UniProt.org.


 French
French Deutsch
Deutsch